📢 转载信息
原文作者:Audra Devoto, Owen Janson, Adam Perry, Christopher Brown, Jamal Arif, and Pavel Novichkov
本文由 Metagenomi 的 Audra Devoto、Owen Janson 和 Christopher Brown,以及 Tennex 的 Adam Perry 撰写。
增强高价值酶的广泛自然多样性的一种有前途的策略是使用生成式 AI,特别是蛋白质语言模型(pLM),通过在已知酶上进行训练,从而生成比现有数量多出几个数量级的给定酶类别的预测示例。通过生成式 AI 扩展自然酶多样性具有许多优势,包括可以获得可能在人体细胞中具有增强的稳定性、特异性或功效的众多酶变体——但根据所用模型的规模和所需酶变体的数量,高通量生成可能会很昂贵。
在 Metagenomi,我们正在使用专有的 CRISPR 基因编辑酶开发潜在的治疗性疗法。我们利用数据库(MGXdb)中广泛的自然酶多样性来识别天然酶候选物,并训练用于生成式 AI 的蛋白质语言模型。使用生成式 AI 扩展天然酶类别使我们能够获得给定酶类别的额外变体,这些变体通过多模型工作流进行过滤,以预测关键的酶特性,并用于支持蛋白质工程活动,以提高酶在给定环境中的性能。
在本文中,我们详细介绍了通过在 AWS Inferentia 上实现 Progen2 模型来降低高通量蛋白质生成式 AI 工作流程成本的方法,这使得在 AWS Batch 和 Amazon Elastic Compute Cloud (Amazon EC2) Spot Instances 上实现酶变体的高通量生成成本降低了高达 56%。这项工作是与 AWS Neuron 团队和 Tennex 的工程师合作完成的。
Progen2 在 AWS Inferentia 上的部署
PyTorch 模型可以使用 AWS Neuron 核心作为加速器,这促使我们使用基于 AWS Inferentia 的 EC2 Inf2 实例类型进行高通量蛋白质设计工作流,以利用其成本效益和作为 Spot 实例的更高可用性。我们选择自回归 Transformer 模型 Progen2 在 EC2 Inf2 实例类型上实现,因为它满足了我们从基于早期在具有 NVIDIA L40S GPU 的 EC2 实例(g6e.xlarge)上运行 Progen2 的微调模型进行高通量合成蛋白质生成的需求,并且 Neuron 对 Transformer 解码器类型模型有既定的支持。为了在 EC2 Inf2 实例上实现 Progen2,我们使用分桶技术追踪了使用专有酶训练的自定义 Progen2 检查点。将模型追踪到多种尺寸并通过生成序列进行优化,即在连续增大的追踪模型上生成序列,将前一个模型的输出作为提示传递给下一个模型,从而最大限度地减少生成每个 token 所需的推理时间。
然而,为使 Progen2 能够在 EC2 Inf2 实例上运行而采用的追踪和分桶方法会引入一些可能影响模型准确性的更改。例如,Progen2-base 训练时没有使用填充 token,但在 EC2 Inf2 实例上运行时必须将填充 token 添加到输入张量中。为了测试我们的追踪和分桶方法的影响,我们将使用 EC2 Inf2 实例上运行的 progen2-base 模型生成的序列的困惑度和序列长度与使用 NVIDIA GPU 上的原生 progen2-base 模型实现生成的序列进行了比较。
为了测试模型,我们为 10 个提示分别生成了 1,000 个蛋白质序列,分别使用追踪和分桶(AWS AI Chip Inferentia)实现和原生(NVIDIA GPU)实现。提示集是通过从 UniprotKB 中抽取 10 个特性良好的蛋白质创建的,并将每个蛋白质截断到前 50 个氨基酸。为了检查生成序列中的异常情况,使用 progen2-base 的原生实现对两个模型生成的所有序列进行前向传递,并计算每个序列的平均困惑度。下图说明了这种方法。
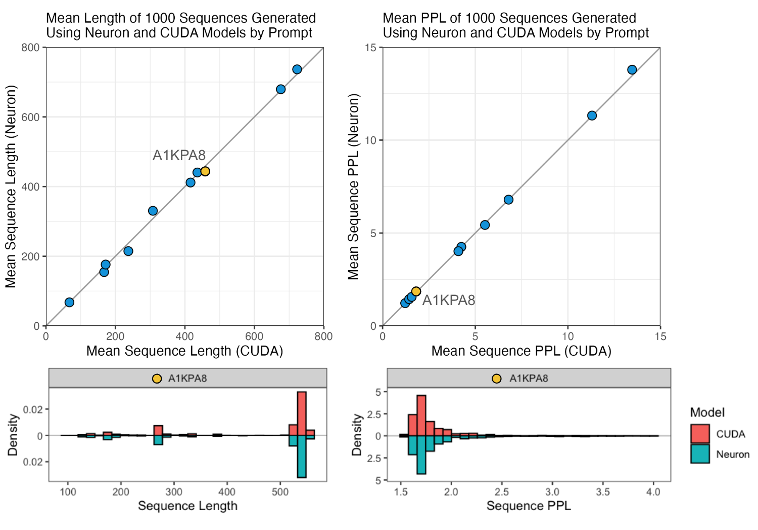
如下图所示,我们发现对于每个提示,使用追踪和分桶实现和原生实现生成的序列的长度和困惑度看起来相似。
AWS Batch 上的规模化推理
在确定了使用 Progen2 在 EC2 Inf2 实例上的基本推理逻辑后,下一步是在大量的计算节点上大规模扩展推理。AWS Batch 是扩展此工作流的理想服务,因为它能够高效地运行数十万个批处理计算作业,并根据提交作业的数量和资源需求,动态配置到最佳数量和类型的计算资源(如 Amazon EC2 按需实例或 Spot 实例)。
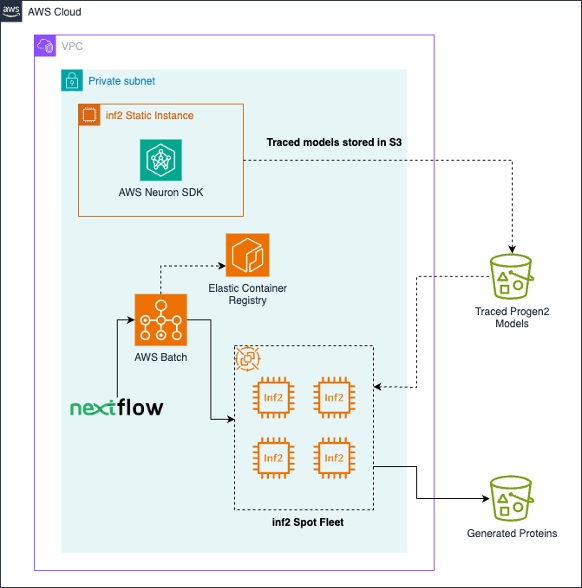
Progen2 按照 最佳实践 作为批处理工作负载实现。作业由用户提交,并在专用的计算环境中运行,该环境协调 Amazon EC2 inf2.xlarge Spot 实例。自定义 Docker 容器存储在 Amazon Amazon Elastic Container Registry (Amazon ECR) 中。模型由每个作业从 Amazon Simple Storage Service (Amazon S3) 中拉取,每个作业结束后,以 FASTA 文件形式生成的序列将放置在 Amazon S3 上。此外,还可以使用 Nextflow 来编排作业、处理自动 Spot 重试以及自动化下游或上游任务。下图说明了解决方案架构。
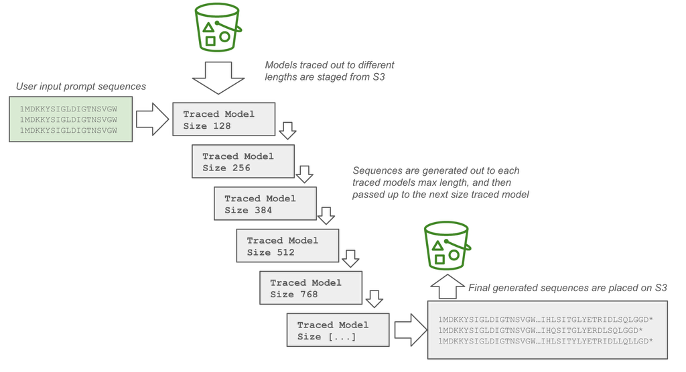
如下图所示,在每个单独的批处理作业中,序列生成过程首先加载可用追踪模型中最小的分桶大小。序列生成到该分桶的最大 token 数,然后丢弃那些已生成停止符或起始符的序列。剩余未完成的序列将被传递给随后更大的分桶尺寸以继续生成,直到达到参数 max_length。最后对张量进行堆叠和解码,并写入写入 Amazon S3 的 FASTA 文件中。
以下是使用 EC2 Inf2 实例设置 AWS Batch 环境的示例配置草图,该环境可以运行 Progen2-neuron 工作负载。
免责声明:这是用于非生产用途的示例配置。在部署之前,您应与您的安全和法务团队合作,以遵守您组织的安全性、监管和合规性要求。
先决条件
在实施此配置之前,请确保已满足以下先决条件:
- 存储在 Amazon ECR 中的 Progen2-NeuronContainerImage
- 一个配置了适当权限的 AWS Identity and Access Management (IAM) 实例角色
- 一个已设置的 Amazon Virtual Private Cloud (Amazon VPC)
{ "LaunchTemplate": { "LaunchTemplateName": "NeuronInfLaunchTemplate", "IamInstanceProfile": "InstanceRole", "BlockDeviceMappings": [ { "DeviceName": "/dev/xvda", "Ebs": { "VolumeSize": 50, "VolumeType": "gp3", "Encrypted": true, "DeleteOnTermination": true } } ], "UserData": { "Base64EncodedScript": [ "#!/bin/bash", "tee /etc/yum.repos.d/neuron.repo > /dev/null <<EOF", "[neuron]", "name=Neuron YUM Repository", "baseurl=https://yum.repos.neuron.amazonaws.com", "enabled=1", "metadata_expire=0", "EOF", "rpm --import https://yum.repos.neuron.amazonaws.com/GPG-PUB-KEY-AMAZON-AWS-NEURON.PUB", "yum install kernel-devel-$(uname -r) kernel-headers-$(uname -r) -y", "yum install unzip -y", "curl 'https://awscli.amazonaws.com/awscli-exe-linux-x86_64.zip' -o awscliv2.zip", "unzip awscliv2.zip", "./aws/install", "rm -rf aws awscliv2.zip", "yum install aws-neuronx-dkms-2.10.11.0 -y", "yum update -y ecs-init", "systemctl restart docker" ] } }, "ComputeEnvironment": { "Name": "NeuronInfComputeEnvironmentSpot", "Type": "MANAGED", "ComputeResources": { "Type": "SPOT", "InstanceTypes": ["inf2.xlarge"], "MaxvCpus": 128, "MinvCpus": 0, "BidPercentage": 20, "AllocationStrategy": "SPOT_CAPACITY_OPTIMIZED", "LaunchTemplate": { "Name": "NeuronInfLaunchTemplate", "Version": "$Latest" }, "InstanceRole": "InstanceRole", "SpotFleetRole": "EC2SpotFleetTaggingRole", "Vpc": "MyVPC" }, }, "JobQueue": { "Name": "NeuronInfSpotJobQueue", "Priority": 10, "ComputeEnvironmentOrder": [ { "ComputeEnvironment": "NeuronInfComputeEnvironmentSpot", "Order": 1 } ] }, "JobDefinition": { "JobDefinitionName": "progen2-neuron-inference", "PlatformCapabilities": ["EC2"], "ContainerOrchestrationType": "ECS", "ContainerProperties": { "Image": "Progen2-NeuronContainerImage", "Command": [ "/opt/generate_sequences.sh", "Ref::S3_ASSETS_PATH", "Ref::S3_INPUT_PROMPT_PATH", "Ref::S3_OUTPUT_PATH", "Ref::MAX_SEQUENCE_LENGTH", "Ref::NUMBER_OF_SEQUENCES", "Ref::TEMPERATURE" ], "ResourceRequirements": [ { "Type": "MEMORY", "Value": "15360" }, { "Type": "VCPU", "Value": "4" } ], "LinuxParameters": { "Devices": [ { "HostPath": "/dev/neuron0", "ContainerPath": "/dev/neuron0", "Permissions": [] } ] }, "ReadonlyRootFilesystem": false }, "Parameters": { "S3_INPUT_PROMPT_PATH": "default prompt s3 path", "MAX_SEQUENCE_LENGTH": "500", "S3_OUTPUT_PATH": "default output path", "TEMPERATURE": "0.5", "S3_ASSETS_PATH": "default model path", "NUMBER_OF_SEQUENCES": "5" } } }下面是容器化 Progen2-neuron 的示例 Dockerfile。该容器镜像基于 Amazon Linux 2023 构建,包含了在 AWS Neuron 上运行 Progen2 所需的组件——包括 Neuron SDK、Python 依赖项、PyTorch 和 Transformers 库。它还为 Hugging Face 操作配置了离线模式,并包含了所需的序列生成脚本。
FROM amazonlinux:2023
# Add the Neuron YUM repository
RUN echo "[neuron]" > /etc/yum.repos.d/neuron.repo && \
echo "name=Neuron YUM Repository" >> /etc/yum.repos.d/neuron.repo && \
echo "baseurl=https://yum.repos.neuron.amazonaws.com" >> /etc/yum.repos.d/neuron.repo && \
echo "enabled=1" >> /etc/yum.repos.d/neuron.repo && \
echo "metadata_expire=0" >> /etc/yum.repos.d/neuron.repo
RUN rpm --import https://yum.repos.neuron.amazonaws.com/GPG-PUB-KEY-AMAZON-AWS-NEURON.PUB \
&& yum install -y \
python3-pip \
aws-neuronx-runtime-lib-2.25.57.0_166c7a468-1 \
aws-neuronx-tools-2.23.9.0-1 \
unzip \
procps-ng
RUN curl "https://awscli.amazonaws.com/awscli-exe-linux-x86_64.zip" -o "awscliv2.zip" \
&& unzip awscliv2.zip \
&& ./aws/install \
&& rm -rf awscliv2.zip aws
RUN pip install \
setuptools \
transformers==4.51.3
RUN pip install \
--no-cache-dir \
--index-url https://pip.repos.neuron.amazonaws.com \
--extra-index-url https://pypi.org/simple \
torch-neuronx===2.1.2.2.3.2 \
torch===2.1.2
ENV PATH="/opt/aws/neuron/bin:${PATH}"
WORKDIR /opt
COPY generate_sequences.py generaterate_sequences.sh ./
# force offline mode for hub and datasets
ENV HF_HUB_OFFLINE=1
ENV HF_DATASETS_OFFLINE=1以下是编排 Progen2 在 EC2 Inf2 实例上进行序列生成工作流的 generate_sequences.sh 示例。
#!/usr/bin/env bash
set -ex
mkdir /scratch
local_assets_directory="/scratch/assets"
local_prompt_file="/scratch/prompt.fasta"
if [ "$#" -ne 6 ]; then echo "Incorrect number of arguments supplied." echo "Usage: $0 <S3_ASSETS_PATH> <S3_INPUT_PROMPT_PATH> <S3_OUTPUT_PATH> <MAX_SEQUENCE_LENGTH> <NUMBER_OF_SEQUENCES> <TEMPERATURE>" exit 1
fi
S3_ASSETS_PATH="$1"
S3_INPUT_PROMPT_PATH="$2"
S3_OUTPUT_PATH="$3"
MAX_SEQUENCE_LENGTH="$4"
NUMBER_OF_SEQUENCES="$5"
TEMPERATURE="$6"
# Download the model, tokenizer, and NEFF files from S3
# For example, if the S3_MODEL_PATH is s3://bucket/job-assets/progen2-base, the files will be in the following paths:
aws s3 sync "$S3_ASSETS_PATH" "$local_assets_directory"
aws s3 cp "$S3_INPUT_PROMPT_PATH" "$local_prompt_file"
# The cached model from S3 expects supporting Python files to be in the ./model directory
ln -s $local_assets_directory/model model
python3 /opt/generate_sequences.py \
--assets_directory $local_assets_directory \
--prompt_file $local_prompt_file \
--number_of_sequences "$NUMBER_OF_SEQUENCES" \
--max_sequence_length "$MAX_SEQUENCE_LENGTH" \
--temperature "$TEMPERATURE"
aws s3 cp generated_sequences.fasta "$S3_OUTPUT_PATH/generated_sequences.fasta"成本比较
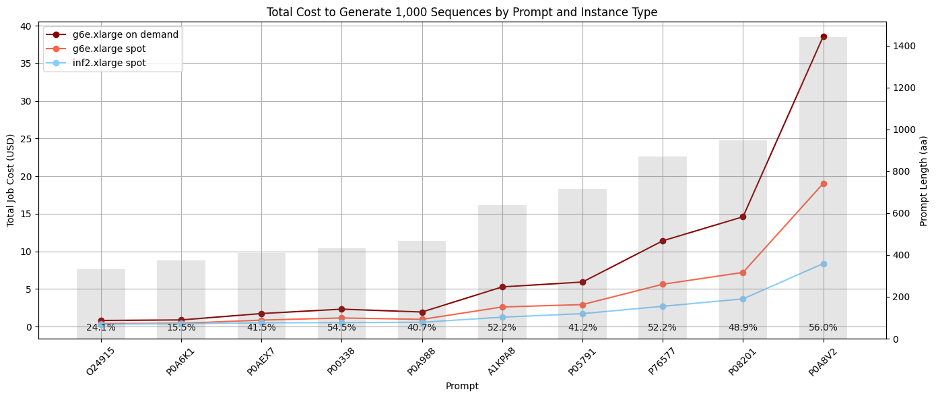
该项目的首要目标是降低使用 Progen2 生成蛋白质序列的成本,以便我们可以利用该模型显著扩展多个酶类别的多样性。为了比较使用两种服务生成序列的成本,我们基于 UniProtKB 中 10 个常见序列的提示生成了 10,000 个序列,温度设置为 1.0。批处理作业并行运行,每个作业为单个提示生成 100 个序列。我们观察到,对于较长的序列,在 Amazon EC2 Inf2 Spot 实例上实现 Progen2 的成本明显低于在 Amazon EC2 G6e Spot 实例上的实现,节省成本高达 56%。这些成本估算包括对由 NVIDIA L40S Tensor Core GPU 驱动的 Amazon EC2 g6e.xlarge 实例预期的 20% 中断频率,以及对 EC2 inf2.xlarge 实例预期的 5% 中断频率(参考 Amazon EC2 Spot 中断频率顾问)。下图说明了总成本,其中灰色条代表生成序列的平均长度。
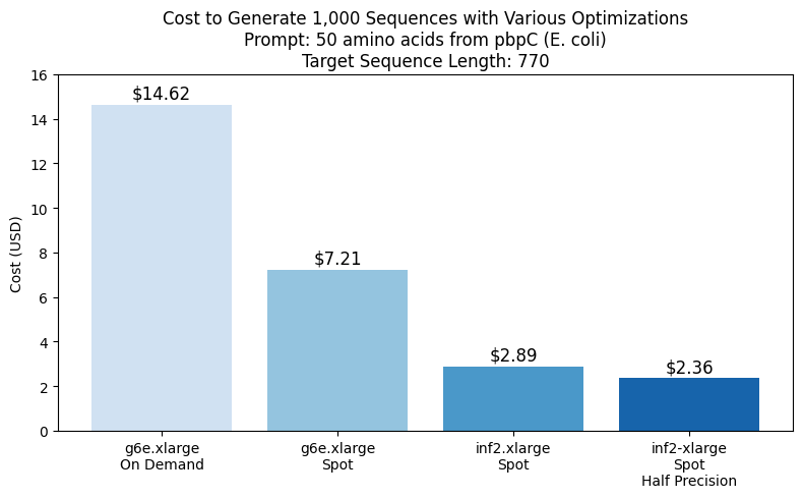
通过以半精度运行作业可以实现额外的成本节省,这似乎产生了等效的结果,如下一图所示。
值得注意的是,由于在生成过程中向序列添加新 token 的时间会以二次方关系扩展,因此生成序列的时间和成本在很大程度上取决于您尝试生成的序列类型。总的来说,我们观察到生成序列的成本取决于所使用的特定模型检查点以及模型生成的序列长度的分布。对于成本估算,我们建议使用您选择的模型生成一小部分酶,然后推断更大生成集的成本,而不是尝试根据先前实验的成本进行计算。
将生成扩展到数百万个蛋白质
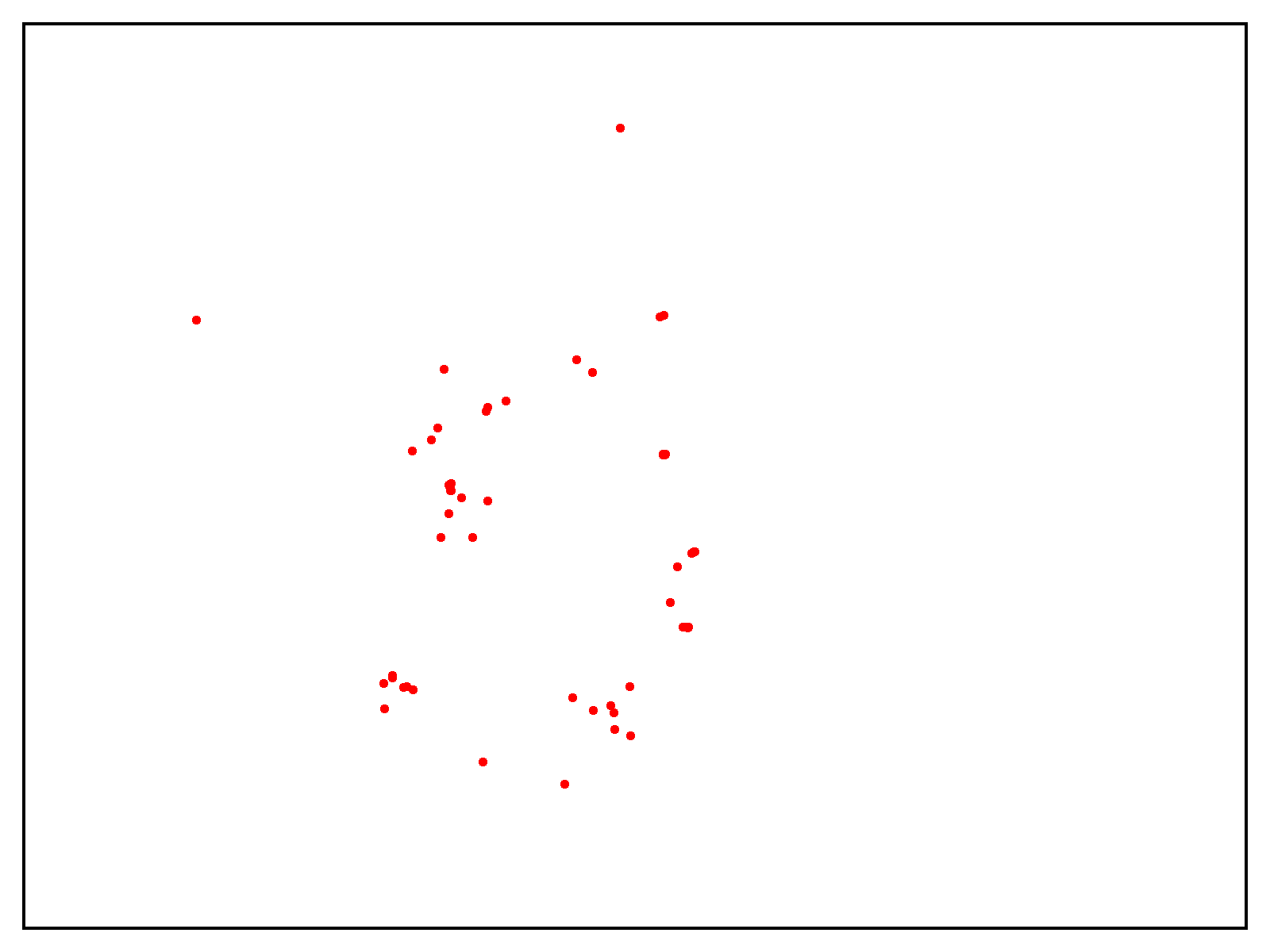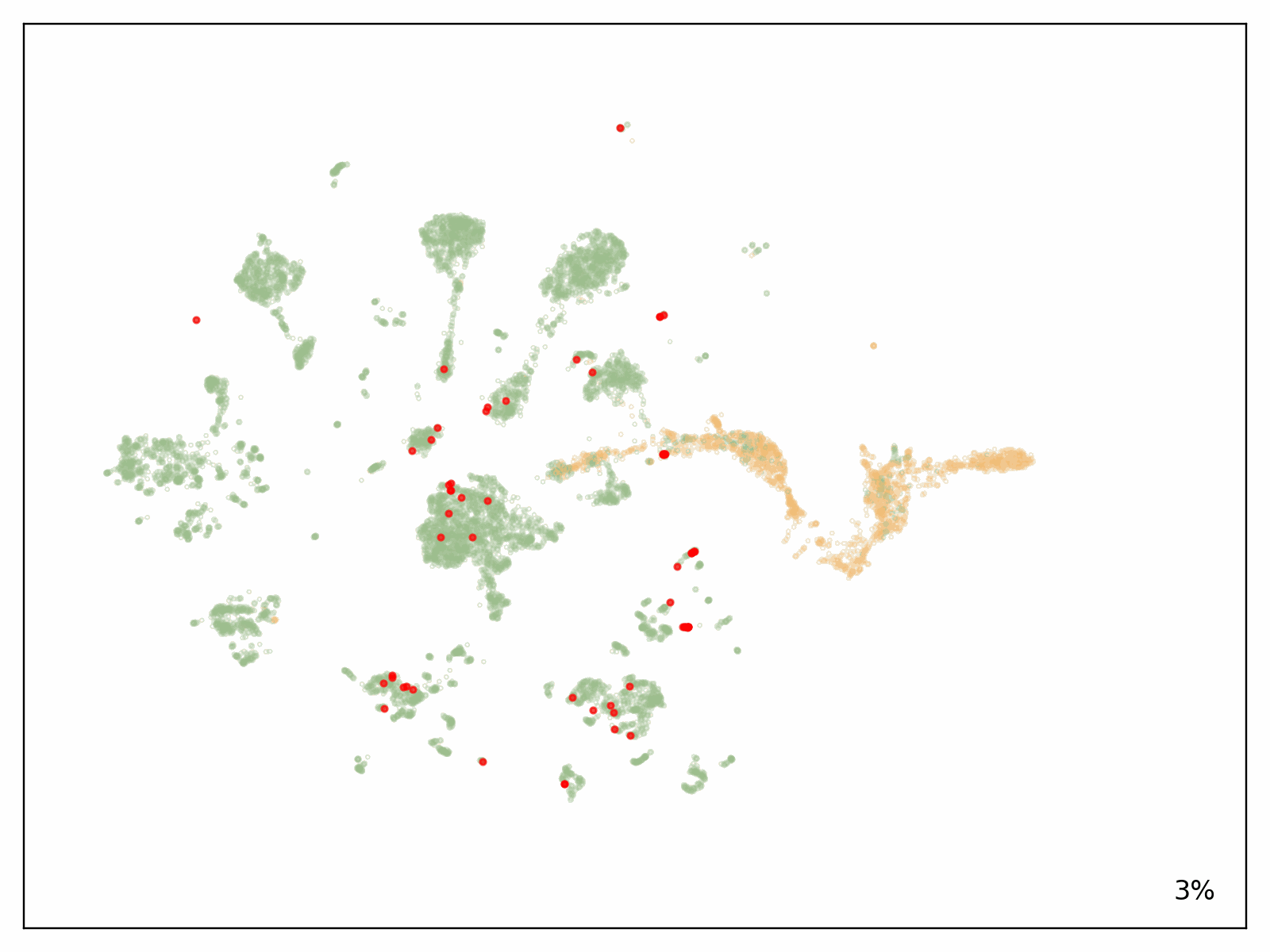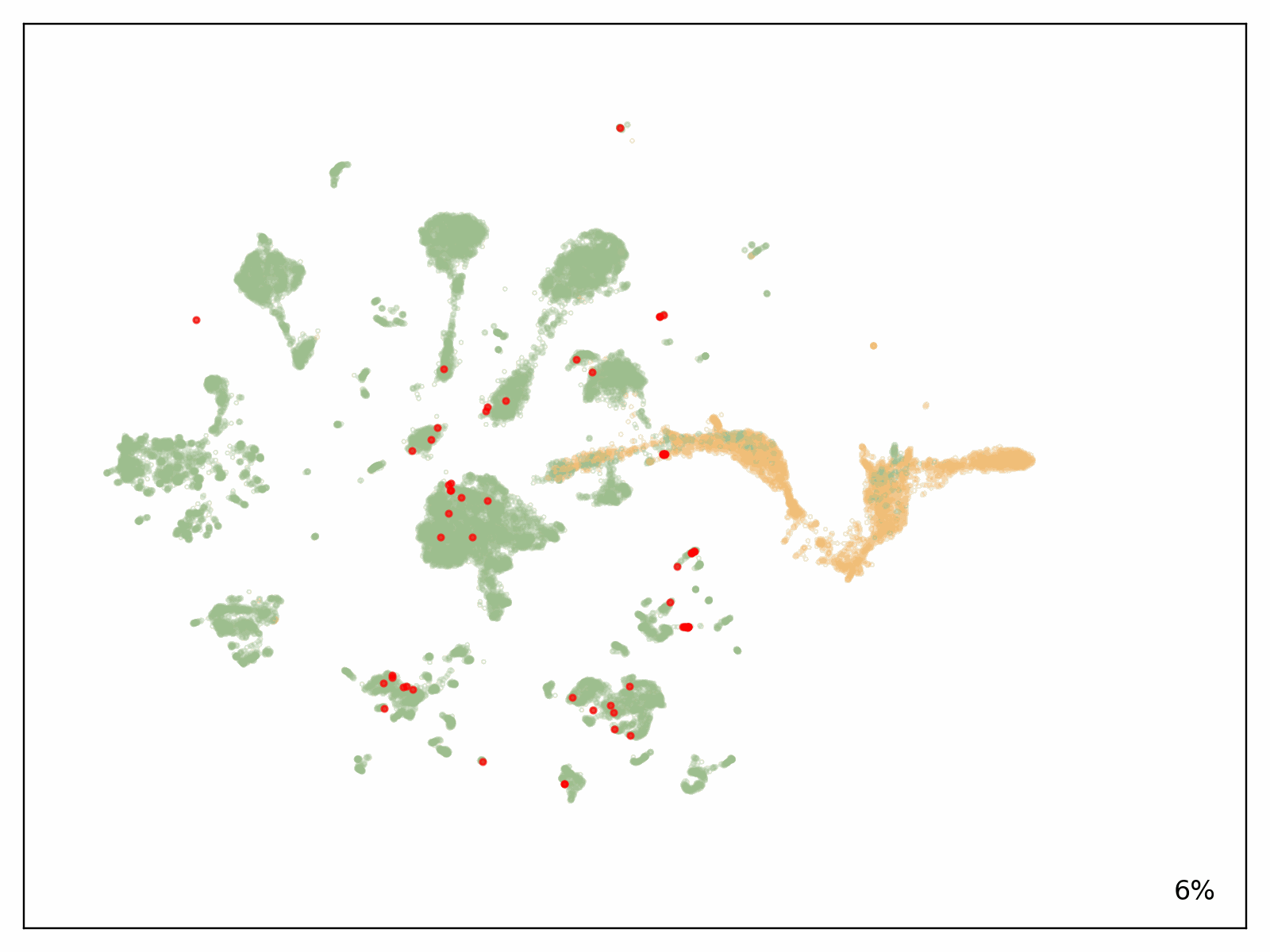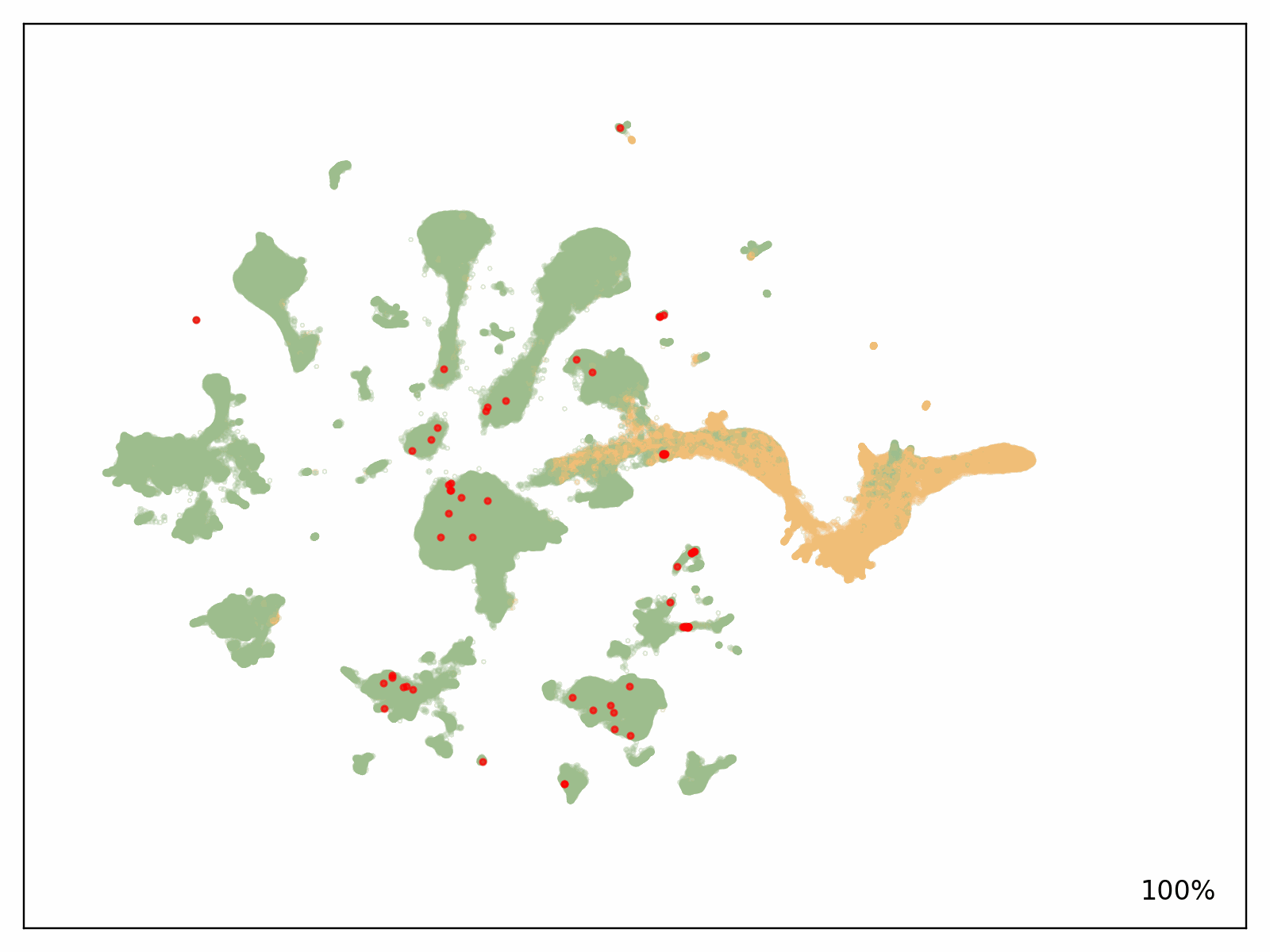
为了测试我们解决方案的可扩展性,Metagenomi 使用 Metagenomi 庞大且专有的宏基因组数据集中有价值但稀有的酶类别的天然示例对模型进行了微调。该模型使用传统 GPU 实例进行微调,然后追踪到 AWS AI 芯片 Inferentia 上。使用我们的批处理和 Nextflow 管道,我们启动了批处理作业,生成了超过 100 万种酶,调整了作业之间的生成参数,以测试不同采样方法、温度和精度的影响。使用我们优化的 AWS AI 管道(包括 EC2 Inf2 Spot 重试产生的成本)生成的总计算成本为 2,613 美元(请参阅前面对估算工作负载成本的注释)。生成序列使用 AI 和传统序列验证技术的组合进行了验证。使用 mmseqs 对序列进行去重,过滤掉适当的长度,使用 hmmsearch 检查正确的结构域,使用 ESMFold 进行折叠,并使用 AMPLIFY_350M 进行嵌入。结构用于与该类别的已知酶进行比较,嵌入用于验证内在酶的适应性。生成结果如下图所示,其中几万个生成式 AI 酶绘制在嵌入空间中。用作提示的天然、表征的酶以红色显示,通过所有过滤的生成式 AI 酶以绿色显示,未通过过滤的生成式 AI 酶以橙色显示。
结论
在本文中,我们概述了通过使用 Amazon EC2 Inf 实例将大型蛋白质设计项目的成本降低高达 56% 的方法,这使得 Metagenomi 能够使用基于我们专有蛋白质数据集训练的模型,跨越多个高价值蛋白质类别生成数百万种新型酶。此实现展示了 AWS Inferentia 如何使大规模蛋白质生成对于生物技术应用更具可访问性和经济性。要了解有关 EC2 Inf 实例的更多信息并开始在 AWS Neuron 上实施您自己的工作流程,请参阅 AWS Neuron 文档。要阅读更多关于 Metagenomi 发现的新型酶的信息,请参阅 Metagenomi 的 出版物和海报。
作者简介
 Audra Devoto 是一位数据科学家,拥有宏基因组学背景,并在 AWS 上处理大型基因组数据集方面拥有多年经验。在 Metagenomi,她构建基础设施以支持大规模分析项目,并促成从 MGXdb 中发现新型酶。
Audra Devoto 是一位数据科学家,拥有宏基因组学背景,并在 AWS 上处理大型基因组数据集方面拥有多年经验。在 Metagenomi,她构建基础设施以支持大规模分析项目,并促成从 MGXdb 中发现新型酶。
 Owen Janson 是 Metagenomi 的生物信息学工程师,专注于构建工具和云基础设施以支持海量基因组数据集的分析。
Owen Janson 是 Metagenomi 的生物信息学工程师,专注于构建工具和云基础设施以支持海量基因组数据集的分析。
 Adam Perry 是一位经验丰富的云架构师,在 AWS 拥有深厚的专业知识,他为数百家企业设计和自动化了复杂的云解决方案。作为 Tennex 的联合创始人,他领导技术战略,构建了定制工具,并与他的团队密切合作,帮助早期生物技术公司在云上安全高效地扩展。
Adam Perry 是一位经验丰富的云架构师,在 AWS 拥有深厚的专业知识,他为数百家企业设计和自动化了复杂的云解决方案。作为 Tennex 的联合创始人,他领导技术战略,构建了定制工具,并与他的团队密切合作,帮助早期生物技术公司在云上安全高效地扩展。
 Christopher Brown 博士是 Metagenomi 发现团队的负责人。他是一位成就卓著的科学家和宏基因组学专家,领导了用于基因编辑应用的众多新型酶系统的发现和表征。
Christopher Brown 博士是 Metagenomi 发现团队的负责人。他是一位成就卓著的科学家和宏基因组学专家,领导了用于基因编辑应用的众多新型酶系统的发现和表征。
 Jamal Arif 是 AWS 的高级解决方案架构师兼生成式 AI 专家,在帮助客户设计和运行下一代 AI 和云原生架构方面拥有十多年的经验。他的工作重点是代理 AI、Kubernetes 和现代化框架,指导企业进行可扩展的采用策略和生产级设计模式。Jamal 构建思想领导力内容,并在 AWS Summit、re:Invent 和行业会议上发表演讲,分享构建安全、有弹性且高影响力 AI 解决方案的最佳实践。
Jamal Arif 是 AWS 的高级解决方案架构师兼生成式 AI 专家,在帮助客户设计和运行下一代 AI 和云原生架构方面拥有十多年的经验。他的工作重点是代理 AI、Kubernetes 和现代化框架,指导企业进行可扩展的采用策略和生产级设计模式。Jamal 构建思想领导力内容,并在 AWS Summit、re:Invent 和行业会议上发表演讲,分享构建安全、有弹性且高影响力 AI 解决方案的最佳实践。
 Pavel Novichkov 博士是 AWS 的高级解决方案架构师,专注于基因组学和生命科学。他拥有超过 15 年的生物信息学和云开发经验,帮助医疗保健和生命科学初创公司在 AWS 上设计和实施基于云的解决方案。他在美国国立生物技术信息中心(NIH)完成了博士后研究,并在伯克利实验室担任计算研究科学家超过 12 年,期间他共同开发了创新的 NGS 技术,该技术被认为是伯克利实验室历史上前 90 项突破之一。
Pavel Novichkov 博士是 AWS 的高级解决方案架构师,专注于基因组学和生命科学。他拥有超过 15 年的生物信息学和云开发经验,帮助医疗保健和生命科学初创公司在 AWS 上设计和实施基于云的解决方案。他在美国国立生物技术信息中心(NIH)完成了博士后研究,并在伯克利实验室担任计算研究科学家超过 12 年,期间他共同开发了创新的 NGS 技术,该技术被认为是伯克利实验室历史上前 90 项突破之一。
🚀 想要体验更好更全面的AI调用?
欢迎使用青云聚合API,约为官网价格的十分之一,支持300+全球最新模型,以及全球各种生图生视频模型,无需翻墙高速稳定,文档丰富,小白也可以简单操作。


评论区