📢 转载信息
原文作者:Edwin Sandanaraj, Charlie Lee, and Hasan Poonawala
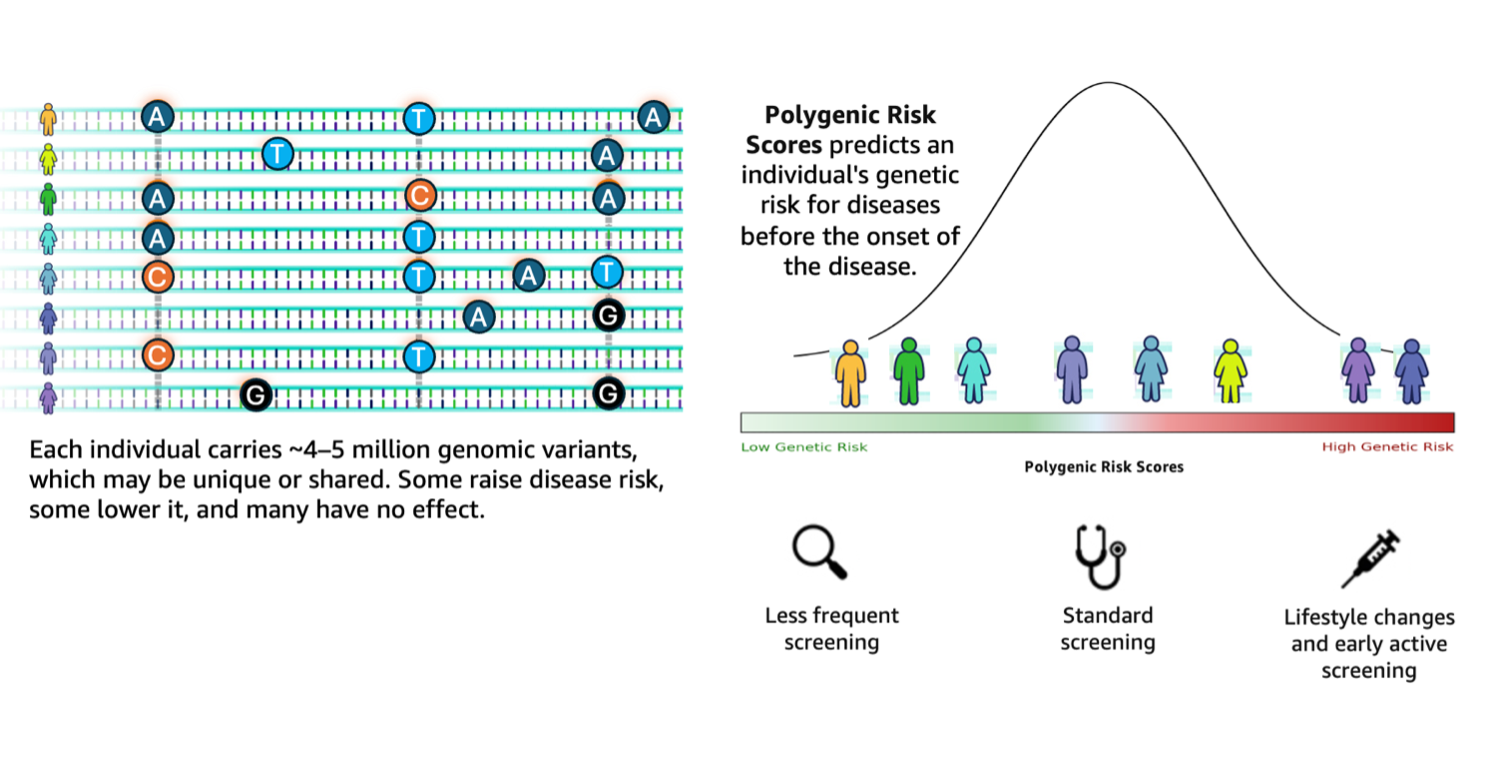
基因组学研究正处于一个变革性的十字路口,测序数据的指数级增长要求同样复杂和精密的分析能力。根据 1000 Genomes Project 的数据,典型的人类基因组在 410 万到 500 万个位点上与参考基因组存在差异,其中大多数变异是单核苷酸多态性(SNP)和短插入缺失(indel)。当这些变异跨个体聚合时,会通过多基因风险评分(PRS)捕获疾病易感性的差异。然而,基因组分析工作流程难以将如此大规模的变异数据转化为可操作的见解。这些流程仍然是分散的,需要研究人员手动协调复杂的管道,包括变异注释、质量过滤以及与 ClinVar 等外部数据库的集成。
AWS HealthOmics 工作流程与 Amazon S3 表和 Amazon Bedrock AgentCore 共同提供了一个变革性的解决方案来应对这些挑战。HealthOmics 工作流程支持将变异等位基因调用格式(VCF)文件的注释与有洞察力的本体论无缝集成。随后,需要将 VEP 注释后的 VCF 文件转换为存储在优化 S3 表中的结构化数据集,以提高跨大型变异队列的查询性能。运行在 Amazon Bedrock AgentCore 上的 Strands Agents SDK 提供了一个安全且可扩展的 AI 代理应用程序,使研究人员无需专业的查询专业知识即可与复杂的基因组数据集进行交互。
在这篇博文中,我们将展示如何利用代理式工作流程,通过自然语言界面,以规模化的方式加速基因组管道的处理和解释。我们演示了一个全面的基因组变异解释代理,它结合了自动化数据处理和智能分析,以解决从原始 VCF 文件摄取到对话式查询界面的整个工作流程。最重要的是,该解决方案消除了传统上将基因组分析限制在专业生物信息学家的技术专业知识障碍。这使得临床研究人员能够上传原始 VCF 文件,并立即提出诸如“哪些患者在 BRCA1 中具有致病性变异?”或“显示此队列中具有耐药性变异的患者”之类的问题。该解决方案的代码可在 AWS 生命科学入门代理的开源工具包存储库中获取。
理解基因组分析中的变异注释
基因组变异解释的基础依赖于全面的注释管道,该管道将原始遗传变异与生物学和临床背景联系起来。变异效应预测器(VEP)和 ClinVar 是现代基因组分析工作流程中两个基本组成部分,它们各自提供研究人员必须集成以获得有意义见解的互补信息。
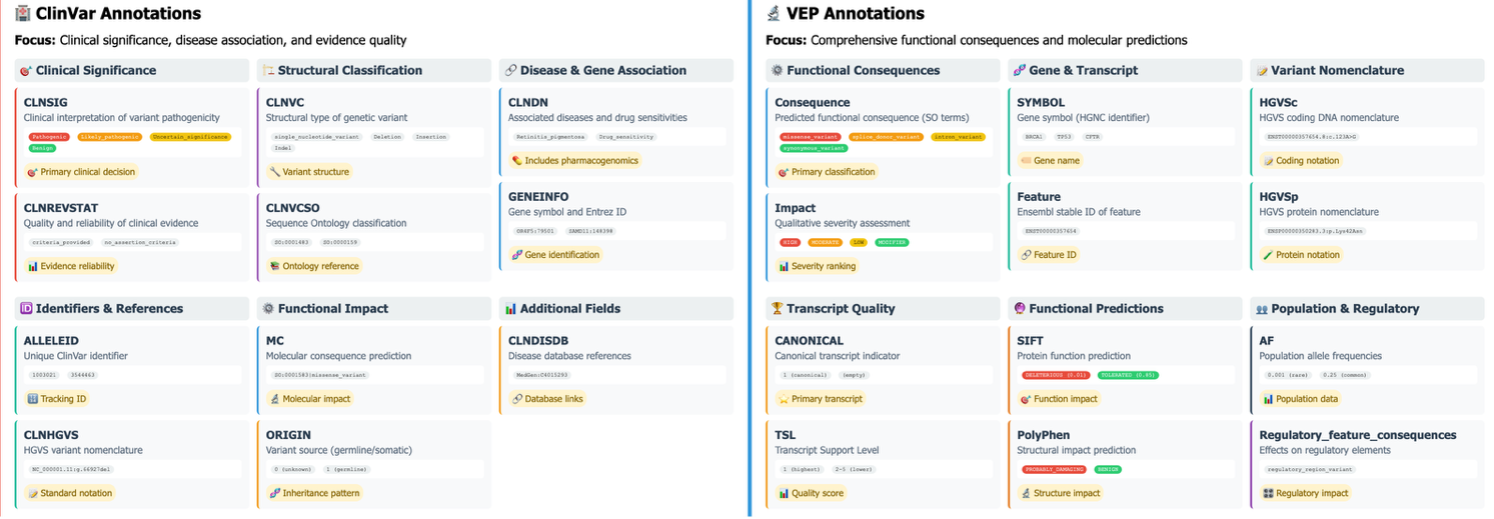
该比较可视化展示了 ClinVar 和 VEP 在基因组变异解释方面具有不同但互补的注释能力。ClinVar 注释(左侧)主要侧重于临床意义评估,直接提供与临床决策相关的已策划的致病性分类(CLNSIG)、证据质量指标(CLNREVSTAT)和疾病关联(CLNDN)。VEP 注释(右侧)提供全面的功能信息,包括后果类型(错义变异、同义变异、内含子变异)、影响严重程度分类(高、中、低、修饰因子)、基因符号以及具有详细位置信息的转录本特异性效应。
当前的注释工作流程挑战
变异注释工作流程通常遵循一个顺序过程,包括:
- 初始 VCF 处理:来自测序系统的原始 VCF 文件需要预处理以规范表示并过滤掉低质量的调用。
- VEP 注释:运行 VEP 工具需要大量的计算资源,特别是对于每个样本包含数百万个变异的全基因组测序数据。根据可用计算资源和注释深度,VEP 分析可能需要 2-8 小时来处理单个基因组。
- ClinVar 集成:必须从 ClinVar 中检索临床注释,并通过单独的过程与变异进行匹配,这需要数据库查找和格式转换。
- 多样本集成:创建队列级分析需要跨样本进行复杂的连接操作,通常使用生成大型、难以高效查询的扁平文件的专用工具来完成。
- 解释:然后,科学家必须使用各种工具来过滤、排序和分析已注释的数据——这个过程通常需要自定义脚本和大量的生物信息学专业知识。这种技术瓶颈意味着临床研究人员无法独立探索其基因组数据,导致从提出生物学问题到获得答案之间延迟数天或数周。
数据集复杂性和规模
基因组变异分析的规模可以通过像 DRAGEN 1000 基因组第三阶段再分析这样的数据集来体现,其中包含:
- 来自不同人群的 2,500 多个单独样本
- 所有样本中大约 8500 万个唯一变异
- 多个注释版本(DRAGEN 3.5、3.7、4.0 和 4.2)需要进行协调
- 复杂的结构变异以及 SNP 和 indel
这种复杂性给依赖于扁平文件处理和手动集成步骤的传统分析管道带来了显著的瓶颈。
解决方案概述
构建基因组队列或计算跨多个患者的 PRS 需要大量的计算资源来使用 VEP 等工具生成联合变异调用表和全面的注释。最关键的是,这些工作流程造成了技术壁垒,只有具备 SQL 专业知识和深入理解变异文件格式的生物信息学家才能提取有意义的见解,使临床研究人员依赖专业的技术团队进行基本的基因组查询。
我们由 AI 驱动的方法的变革性优势在于通过自然语言交互实现基因组分析的民主化。虽然传统的 VEP 管道需要数天的技术专业知识才能回答诸如“哪些患者在药物耐药基因中具有高影响变异?”之类的临床问题,但通过我们的解决方案,研究人员可以以对话的方式提出这些问题,并在几分钟内获得答案。这代表着从技术依赖到自助服务基因组见解的转变,使临床研究人员、肿瘤委员会和基因组学团队无需等待生物信息学支持即可直接探索他们的数据。
我们的解决方案展示了一个由生成式 AI 驱动的基因组变异解释代理,它结合了自动化数据处理和智能自然语言分析。该架构解决了整个基因组分析工作流程,从原始 VCF 文件摄取到对话查询界面。
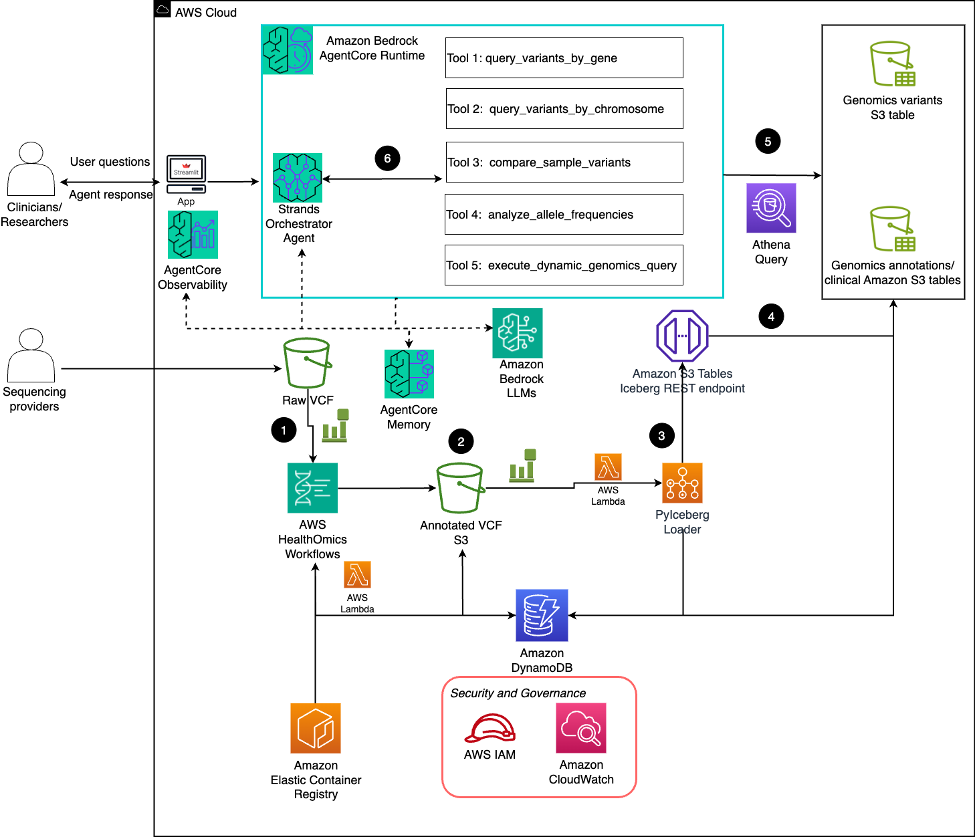
该解决方案遵循六个关键步骤,将原始基因组数据转化为可操作的见解:
- 原始 VCF 处理:来自测序提供商的原始 VCF 文件上传到 Amazon S3 存储,并通过 S3 事件通知触发 AWS Lambda 函数,后者协调 AWS HealthOmics 工作流程。
- VEP 注释:AWS HealthOmics 工作流程使用 VEP 自动处理原始 VCF 文件,在将注释后的结果存储回 S3 之前,并行丰富变异的功能预测和临床注释。
- 事件协调:Amazon EventBridge 监控工作流程完成情况,并触发 Lambda 函数,这些函数在 Amazon DynamoDB 中更新作业状态,而 AWS Batch Fargate 计算环境将 VEP 注释后的 VCF 文件和 ClinVar 注释转换为 Iceberg 格式(使用 PyIceberg 模块)。
- 数据组织:PyIceberg 加载器与 Amazon S3 表 Iceberg Rest Endpoint 交互。Amazon S3 表在 AWS Glue Data Catalog 中注册表元数据。架构信息(列、数据类型、分区)被编入目录,用于注释后的 VCF 和 ClinVar 注释。它还为下游分析建立了分析连接器。
- SQL 驱动的分析:Amazon Athena 通过列式存储格式提供对基因组数据的 SQL 查询功能,使得能够对数百万个变异进行大规模分析,并获得理想的查询响应。
- 自然语言交互:由 Amazon Bedrock LLM 在 AgentCore Runtime 上支持的 Strands 编排代理,通过五个专门执行 Athena 查询的工具提供自然语言界面:
- query_variants_by_gene:检索与特定基因相关的变异
- query_variants_by_chromosome:促进特定于染色体的变异分析
- compare_sample_variants:支持跨患者样本的比较基因组学
- analyze_allele_frequencies:提供人群遗传学见解
- execute_dynamic_genomics_query:支持灵活的、即席的分析请求
该架构通过 AWS IAM 包含全面的安全控制,用于细粒度访问管理,并通过 Amazon CloudWatch 进行监控。自动化的、事件驱动的管道支持 VCF 文件的大规模并行处理,该管道自动适应不断增长的基因组数据集,同时保持一致的注释质量和分析能力。
Amazon S3 表与 PyIceberg:将 VCF 转化为结构化队列
Amazon S3 表与 PyIceberg 合作,将 VEP 注释后的 VCF 文件转换为结构化队列,即针对 AI 驱动分析进行优化的可查询数据集。这为自然语言界面高效交互复杂基因组数据创建了数据基础。
PyIceberg 在 S3 表格式中创建 Apache Iceberg 表,提供以下优势:
- 最佳查询:代理可以通过优化的列式存储,以最少的延迟对数百万个变异执行复杂的基因组查询,将以前需要数小时 SQL 开发和执行的分析转化为即时的对话响应。
- 丰富的注释访问:VEP 和 ClinVar 注释通过 Amazon Athena 变得可通过 SQL 直接查询,允许 AI 代理提取特定的基因组见解。
- 队列级分析:结构化 Iceberg 格式(PyIceberg)通过自然语言支持跨患者队列的高效比较,用于人群级别查询。
在 S3 表中将变异数据与注释数据分离,为 AI 驱动的分析创建了理想的基础,因为基因组变异 S3 表包含代理可以快速过滤的核心位置信息,而注释/临床 S3 表则包含解释所需的丰富功能和临床背景。通过这种结构,Strands 代理可以通过 AWS Glue Data Catalog 连接器构建目标查询,以精确回答用户的问题。
这种从原始 VCF 文件到结构化表的转换使得研究人员能够通过 Strands 编排代理 [KM1] 在 Amazon Bedrock AgentCore 上以对话方式查询复杂的基因组数据集。
利用 Strands Agents 和 AgentCore Runtime 进行智能基因组分析
对话界面是我们基因组 AI 解决方案的核心创新,它是使用 Strands Agents SDK 构建并在 Amazon Bedrock AgentCore Runtime 上部署的。这个复杂的 AI 代理理解复杂的基因组概念,并将自然语言查询转化为针对结构化基因组数据集的适当分析操作。
AgentCore Runtime 是一个专为部署和扩展动态 AI 代理和工具而设计的安全、无服务器运行时。该解决方案为基因组分析提供了几项关键优势:
- 模型和框架灵活性:AgentCore 服务是可组合的,并且可以与 Amazon Bedrock 内部和外部的开源或自定义框架和模型一起工作。
- 多小时代理工作负载:支持长达 8 小时和高达 100MB 有效载荷的长时间运行工作负载。
- 安全性:为每次用户会话提供专用微虚拟机,实现完全隔离。
- 企业级集成:通过 AgentCore Identity 与 AWS IAM 内置身份验证。
- 可观察性:对代理推理和工具调用的全面跟踪。
- 私有资源访问:连接到 Amazon Virtual Private Cloud 内部的数据库和 API。
- 更快的上市时间:AI 代理解决方案的部署和开发周期加速。
有关 Amazon Bedrock AgentCore 功能的详细信息,请参阅 Amazon Bedrock AgentCore 文档。
Strands Agents 通过一种模型驱动的方法为构建具有专业能力的领域特定 AI 代理提供了稳固的基础,该方法使用代理循环概念来协调基因组分析工具。这种迭代推理框架使代理能够根据分析要求动态选择和执行适当的工具。我们的基因组变异解释器实现了五个关键工具,这些工具利用 Amazon S3 表创建的结构化数据:
- 变异查询:将基于基因的问题转化为精确的 Athena SQL 查询,以检索相关的变异。
- 染色体分析:通过自然语言实现区域特定的基因组查询。
- 样本比较:在不需要 SQL 连接的情况下促进跨患者基因组分析。
- 人群频率分析:将发现与 1000 基因组等参考数据集进行情境化。
- 动态查询生成:将复杂的自然语言请求转换为优化的 SQL。
自然语言查询
该代理在处理不同类型的查询方面表现出卓越的能力。在传统模型中,临床研究人员必须等待生物信息学团队编写自定义脚本并运行复杂的分析。研究人员不再需要花费数天时间来设计 SQL 查询和处理 VCF 文件格式,现在可以像与基因组学专家进行对话一样自然地探索他们的基因组数据。
队列级分析
用户:“请以表格形式总结此队列中每位患者的总变异数和致病性?”
对于此查询,代理:
- 使用 execute_dynamic_genomics_query 工具。
- 分析队列样本中的变异数据。
- 生成包含患者计数和变异统计数据的综合队列摘要。
- 以结构化和表格化的摘要格式呈现结果。
队列级频率分析
用户:“请提供此队列中共享的致病性或可能致病性变异的等位基因频率以及 1000 基因组的数据?”
代理将其转换为以下查询:
- 通过运行 execute_dynamic_genomics_query 和 analyze_allele_frequencies 工具,检索患者的致病性变异列表。
- 过滤出临床相关的致病性变异。
- 从 ClinVar 中提取疾病水平信息,从 VEP 中提取等位基因频率。
- 提供带有相关背景信息的结果。
合并症风险关联
用户:“哪些患者在染色体 10:111079820 处的 ADRA2A 基因中存在变异,并且这些患者是否具有任何与他汀类药物或胰岛素抵抗相关的其他高影响变异?”
对于此查询,代理:
- 搜索特定疾病背景下耐药途径中的其他风险变异。
- 在个体患者层面连接临床意义,以确定合并症。
- 提供联合临床和耐药途径的临床意义。
这种自然语言界面最大限度地减少了研究人员掌握复杂 SQL 语法或理解底层数据结构的需求,从而使所有技术背景的临床和研究团队都能民主化地获取基因组见解。
高级分析处理
除了查询之外,基因组变异解释代理还展示了超越基本变异识别的高级分析能力。研究人员可以探索传统上需要数天分析的复杂问题。
临床决策支持
用户:“对患者 NA21144 进行全面分析,并为该患者提供风险分层。”
对于此查询,代理:
- 分析疾病通路基因、药物基因组学中的变异,并提供循证建议。
- 通过结合变异影响预测和临床意义分类来进行风险分层。
- 识别意义未明的变异。
- 标记临床相关基因中的高影响变异。
药物基因组学指导给药策略
研究人员可以利用该代理通过诸如以下查询,对大型队列进行复杂的药物基因组学通路分析:
用户:“在此患者队列中,哪些主要的药物相关通路因遗传变异而显著富集?请为我提供最具影响力的药物基因组学通路和相关患者 ID。”
这使得研究人员可以通过对话界面探索变异频率分布、后果类型模式和基因水平的变异负荷,而无需复杂的 SQL 或生物信息学管道。
优势和局限性
该解决方案有助于解决当前的挑战:
| 挑战 | 解决方案 |
| 初始 VCF 处理 – 低质量调用 | 在做出变异解释决策之前,代理会自动预先检查变异的质量调用。 |
| 大规模 VEP 注释 | 该解决方案使用正确的计算资源以批处理方式(20个一组)自动化 VCF 注释,以实现适当的性能。 |
| ClinVar 集成 | 代理评估查询上下文,并将动态构建联合查询,具体取决于用户的兴趣。 |
| 多样本集成 | Amazon S3 表以 Iceberg 格式集成,使得 VCF 文件队列能够以理想的性能进行查询。 |
| 基因组解释 | 代理理解上下文和用户兴趣,根据注释和内部信息仔细推理,做出知情的决定。 |
该解决方案存在以下限制:
- Lambda 运行时限制:当前的实现使用 AWS Lambda 进行 VCF/GVCF 处理,其最长执行时间为 15 分钟。此限制可能不足以加载大型 VCF 文件或特别大的 GVCF 文件到 Iceberg S3 表中,因为这些操作可能比 Lambda 超时限制花费更长的时间。对于具有大型基因组数据集的生产工作负载,请考虑使用 AWS HealthOmics 工作流程、AWS Batch、ECS 任务或具有更长执行时间的 EC2 实例来处理数据加载过程。
- 架构优化权衡:架构实现使用样本和染色体分区,这针对患者级别的分析进行了优化。然而,队列级别的分析通常需要不同的分区策略和架构设计,才能在大规模时实现最佳性能。在单个架构中同时实现患者级别和队列级别的分析性能,随着队列规模超过数百个样本而变得越来越具有挑战性。对于大规模队列研究(数千到数万个样本),请考虑实施针对特定分析模式优化的单独架构或物化视图,或探索更好地支持人群级别查询的反范式结构。
未来技术演进
该解决方案的模块化架构为 AI 驱动的基因组分析的持续创新奠定了基础。未来的版本可以集成额外的注释数据库、外部 API,并支持多模态分析,将基因组数据与临床记录和影像相结合。针对基因组数据的领域特定微调可以进一步提高解释准确性,而与电子健康记录的集成将提供临点基因组见解。
一个特别有前景的方向是制药研发中的多代理协作,其中这个基因组变异解释代理可以与药物分析、靶点识别、文献证据和假设生成的专业代理协同工作。这种协作代理框架可以通过将变异级别见解直接与治疗开发联系起来,从而显著加速药物发现管道,简化从遗传发现到临床应用的转化过程。
结论
这种下一代基因组代理式 AI 解决方案代表了研究人员和临床医生与基因组数据交互方式的根本转变。通过无缝集成 AWS HealthOmics 进行自动化变异注释和数据转换,以及使用 Amazon Bedrock AgentCore 进行智能解释,我们创建了一个全面的解决方案,解决了整个基因组分析工作流程。
自动化 VEP 注释工作流程、使用 S3 表将 VCF 数据转换为可查询的 Iceberg 表,以及使用 Amazon Bedrock AgentCore 上的 Strands Agents 进行自然语言交互的组合,创建了一个系统,最大限度地减少了变异注释、数据处理和临床解释之间的传统障碍。通过自动化复杂的技术流程并提供直观的交互方法,研究人员现在可以专注于生物学问题,而不是技术实现细节。
随着基因组数据的持续指数级增长和临床应用的日益复杂,像这样的系统将成为推进精准医疗和加速科学发现的基本基础设施。使用 1000 基因组第三阶段再分析数据集展示的解决方案表明,即使是大型基因组队列也可以通过简单的对话界面进行分析,从而实现了高级基因组见解的民主化访问。
该解决方案的代码可在生命科学代理工具包上获取,我们鼓励您探索和构建此模板。有关开始使用 Amazon Bedrock AgentCore 的示例,请查看 Amazon Bedrock AgentCore 存储库。
关于作者
 Edwin Sandanaraj 是 AWS 的一名基因组学解决方案架构师。他拥有神经肿瘤学博士学位和超过 20 年的医疗保健基因组学数据管理和分析经验,为加速亚太及日本地区的精准基因组学工作带来了丰富的知识。他对临床基因组学和多组学在利用云解决方案加速精准医疗方面抱有浓厚的兴趣。
Edwin Sandanaraj 是 AWS 的一名基因组学解决方案架构师。他拥有神经肿瘤学博士学位和超过 20 年的医疗保健基因组学数据管理和分析经验,为加速亚太及日本地区的精准基因组学工作带来了丰富的知识。他对临床基因组学和多组学在利用云解决方案加速精准医疗方面抱有浓厚的兴趣。
 Hasan Poonawala 是 AWS 的高级 AI/ML 解决方案架构师,与医疗保健和生命科学客户合作。Hasan 帮助在 AWS 上设计、部署和扩展生成式 AI 和机器学习应用程序。他在云计算领域的机器学习、软件开发和数据科学方面拥有超过 15 年的综合工作经验。在业余时间,Hasan 喜欢探索自然并与朋友和家人共度时光。
Hasan Poonawala 是 AWS 的高级 AI/ML 解决方案架构师,与医疗保健和生命科学客户合作。Hasan 帮助在 AWS 上设计、部署和扩展生成式 AI 和机器学习应用程序。他在云计算领域的机器学习、软件开发和数据科学方面拥有超过 15 年的综合工作经验。在业余时间,Hasan 喜欢探索自然并与朋友和家人共度时光。
 Charlie Lee 是 AWS 亚太及日本地区的基因组学行业负责人,拥有计算机科学博士学位,专攻生物信息学。作为一位在生物信息学、基因组学和分子诊断领域拥有二十多年经验的行业领导者,他热衷于通过尖端的测序技术加速研究和改善医疗保健...... [内容被截断]
Charlie Lee 是 AWS 亚太及日本地区的基因组学行业负责人,拥有计算机科学博士学位,专攻生物信息学。作为一位在生物信息学、基因组学和分子诊断领域拥有二十多年经验的行业领导者,他热衷于通过尖端的测序技术加速研究和改善医疗保健...... [内容被截断]
🚀 想要体验更好更全面的AI调用?
欢迎使用青云聚合API,约为官网价格的十分之一,支持300+全球最新模型,以及全球各种生图生视频模型,无需翻墙高速稳定,文档丰富,小白也可以简单操作。


评论区