📢 转载信息
原文链接:http://bair.berkeley.edu/blog/2025/04/08/plaid/
原文作者:BAIR Blog
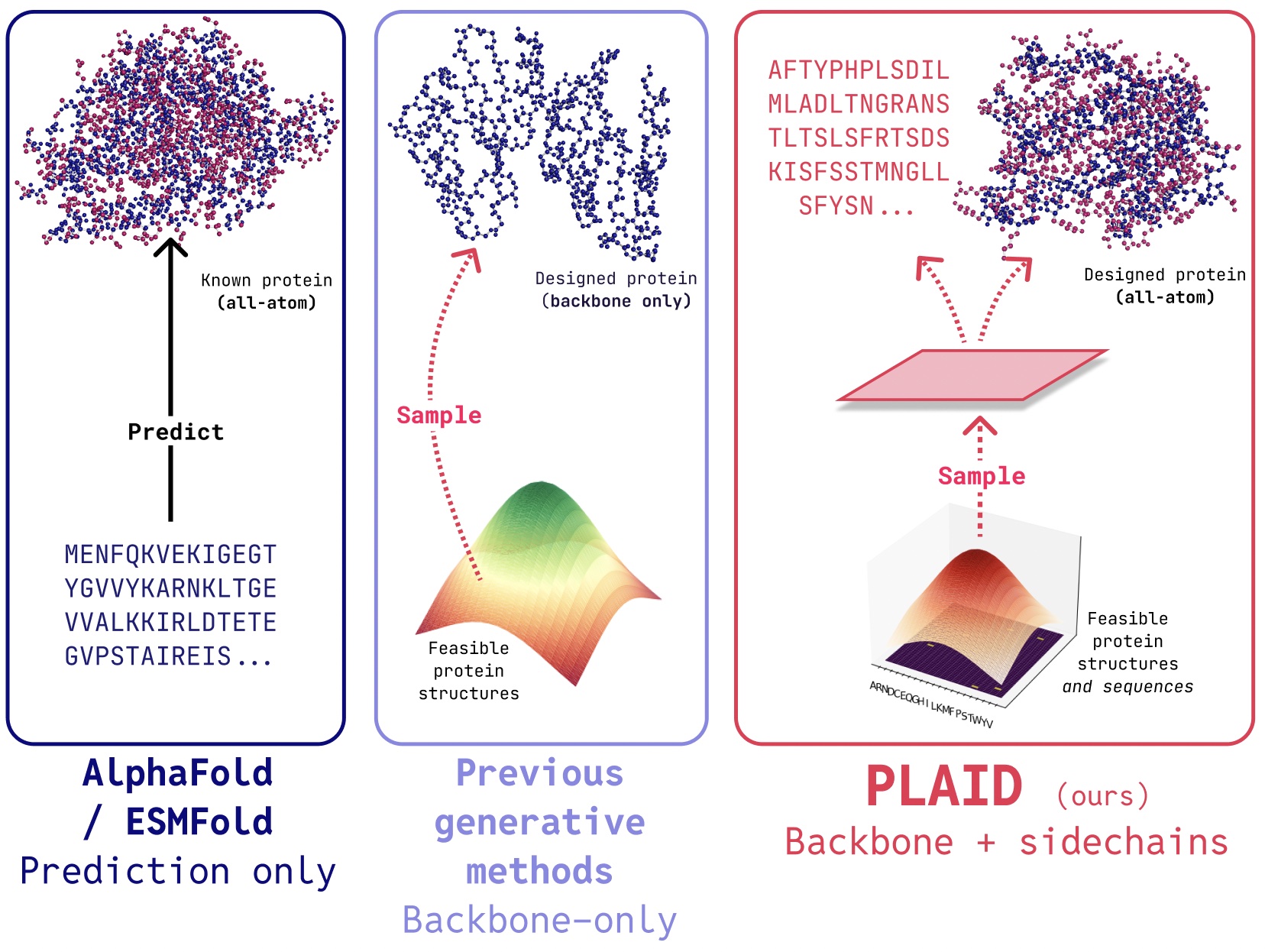
PLAID 是一种多模态生成模型,它通过学习蛋白质折叠模型的潜在空间,可以同时生成蛋白质的 1D 序列和 3D 结构。
2024年诺贝尔奖授予AlphaFold2,标志着AI在生物学中作用得到了重要认可。那么,在蛋白质折叠之后,下一步是什么呢?
在PLAID中,我们开发了一种方法,它学会了从蛋白质折叠模型的潜在空间中采样,以生成新的蛋白质。它可以接受组合式的功能和生物体提示,并且可以在序列数据库上进行训练,这些数据库的规模比结构数据库大 2 到 4 个数量级。与许多早期的蛋白质结构生成模型不同,PLAID 解决了多模态共生成问题设置:同时生成离散的序列和连续的全原子结构坐标。
从结构预测到现实世界的药物设计
尽管最近的工作展示了扩散模型生成蛋白质的潜力,但现有模型仍存在一些限制,使其在现实世界的应用中不切实际,例如:
- 全原子生成:许多现有的生成模型仅产生骨架原子。要生成全原子结构并放置侧链原子,我们需要知道序列。这构成了一个需要同时生成离散和连续模态的多模态生成问题。
- 生物体特异性:用于人类用途的生物制剂需要人性化,以避免被人类免疫系统破坏。
- 控制规范:药物发现并将其交到患者手中是一个复杂的过程。我们如何指定这些复杂的约束呢?例如,即使解决了生物学问题,您也可能决定药片比小瓶更容易运输,这就增加了对溶解性的新约束。
生成“有用”的蛋白质
仅仅生成蛋白质不如控制生成以获得有用的蛋白质那么有价值。这样的接口会是什么样子呢?

作为参考,让我们考虑如何通过组合式文本提示来控制图像生成(引自Liu 等人, 2022的例子)。
在 PLAID 中,我们为控制规范镜像了这种接口。最终目标是通过文本接口完全控制生成,但在这里我们考虑了两个轴的组合约束作为概念验证:功能和生物体:
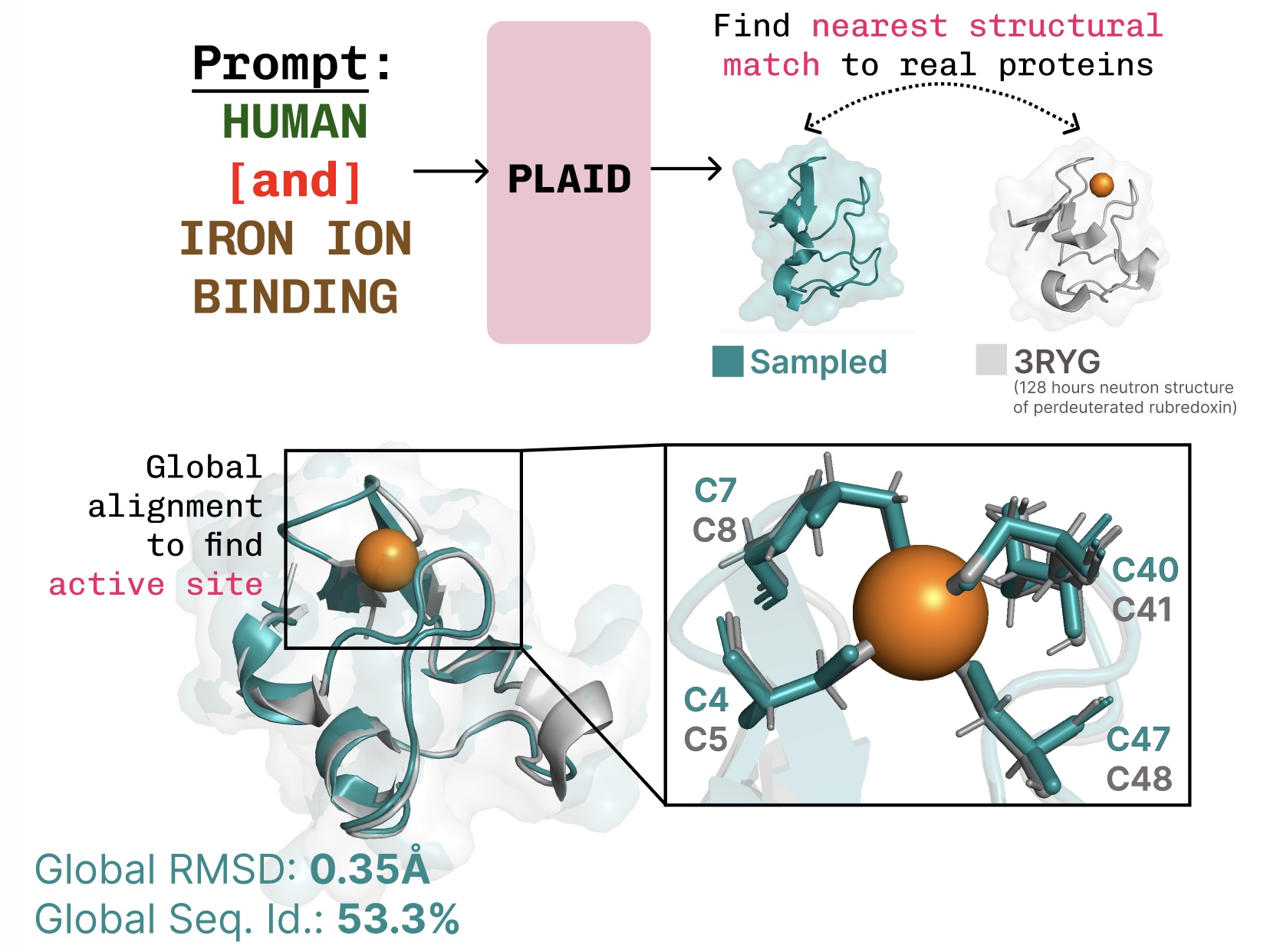
学习功能-结构-序列的联系。 PLAID 学习了金属蛋白中常见的四面体半胱氨酸-Fe2+/Fe3+配位模式,同时保持了高序列多样性。
使用仅序列的训练数据进行训练
PLAID 模型另一个重要方面是,我们只需要序列来训练生成模型! 生成模型学习由其训练数据定义的分布,而序列数据库比结构数据库大得多,因为与实验结构相比,序列更容易获得。
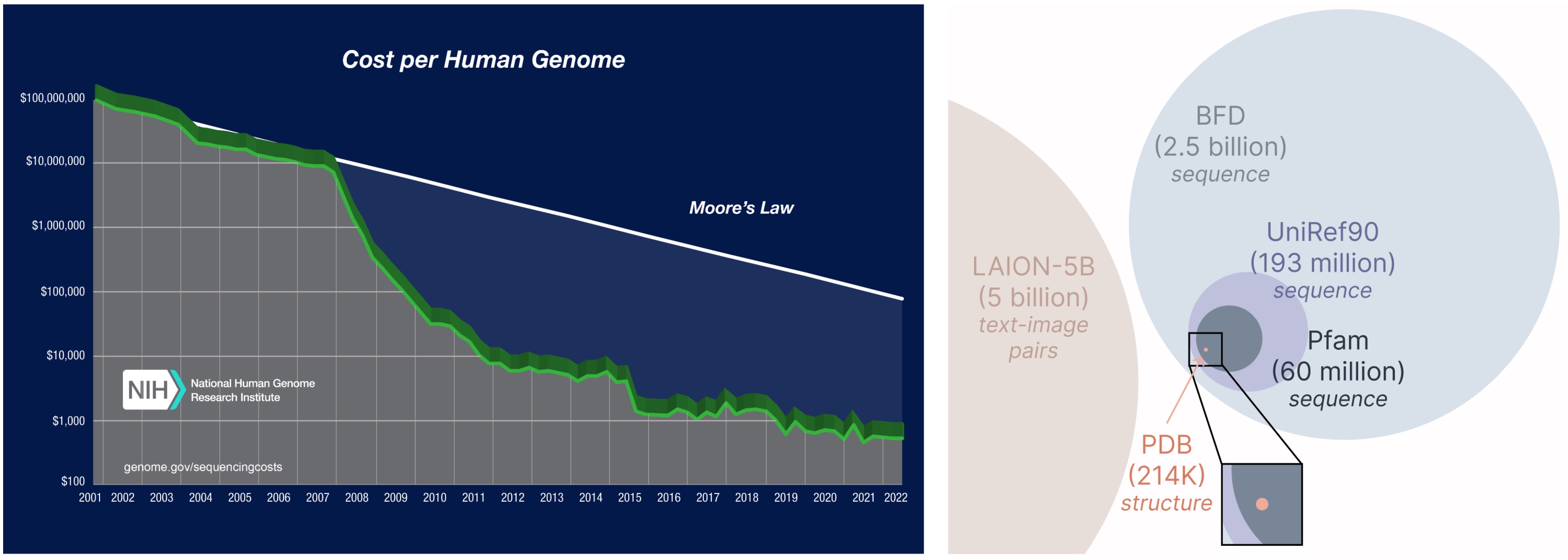
从更大、更广泛的数据库中学习。 获取蛋白质序列的成本远低于实验表征结构,且序列数据库比结构数据库大 2 到 4 个数量级。
它是如何工作的?
我们能够仅使用序列数据训练生成模型来生成结构的原因是,我们学习了蛋白质折叠模型的潜在空间上的扩散模型。然后在推理过程中,在从有效蛋白质的潜在空间采样后,我们可以从蛋白质折叠模型中采用冻结的权重来解码结构。在这里,我们使用 ESMFold,它是 AlphaFold2 模型的后继者,它使用蛋白质语言模型来替代检索步骤。
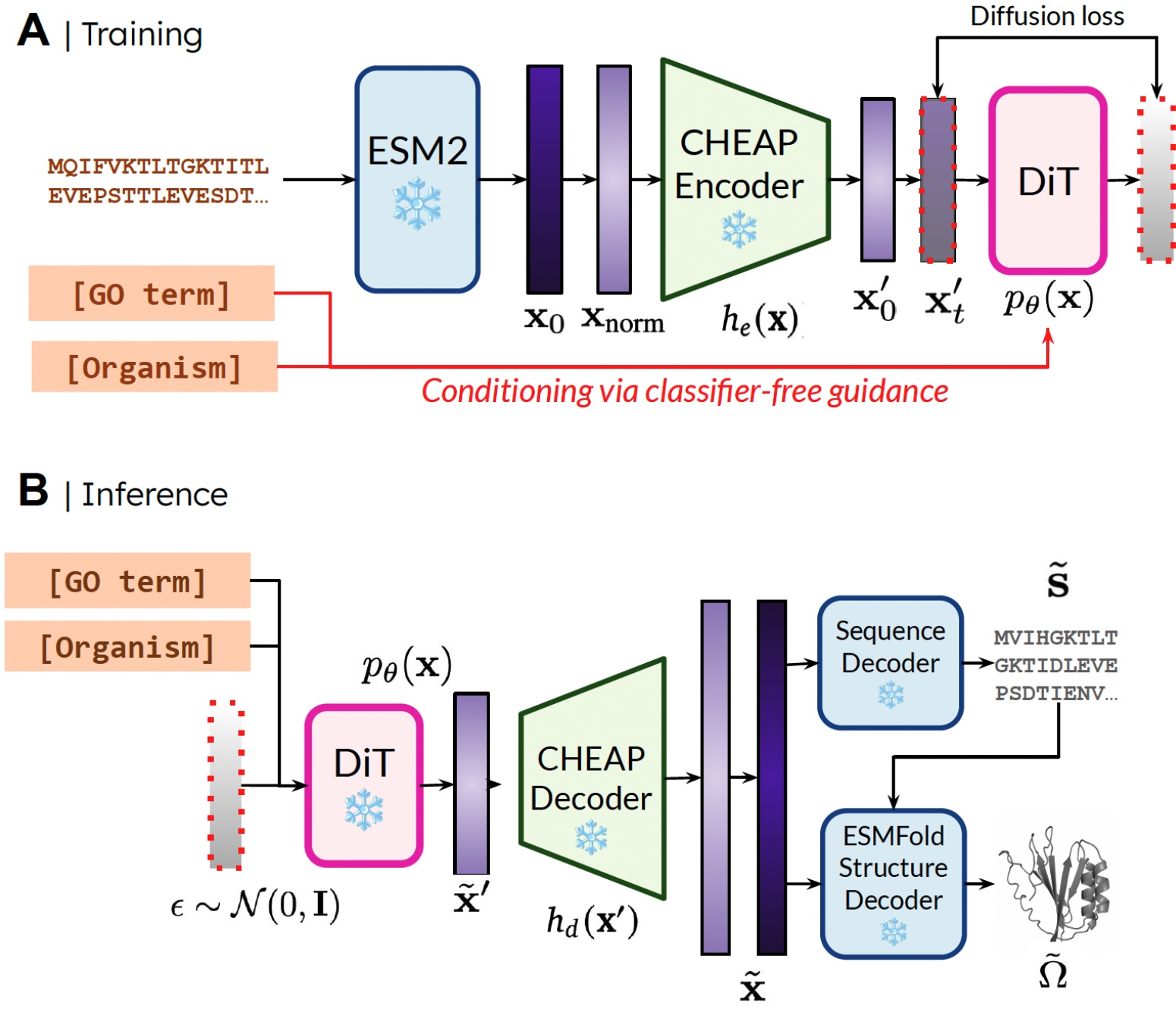
我们的方法。 在训练期间,只需要序列来获得嵌入;在推理期间,我们可以从采样的嵌入中解码序列和结构。❄️ 表示冻结的权重。
通过这种方式,我们可以利用预训练蛋白质折叠模型中包含的结构理解信息来完成蛋白质设计任务。这类似于机器人学中的视觉-语言-动作 (VLA) 模型如何利用在互联网规模数据上训练的视觉-语言模型 (VLM) 中包含的先验知识,以提供感知、推理和理解信息。
压缩蛋白质折叠模型的潜在空间
直接应用此方法的细微之处在于 ESMFold 的潜在空间——事实上,许多基于 Transformer 的模型的潜在空间——需要大量的正则化。这个空间也非常大,因此学习这个嵌入最终会映射到高分辨率图像合成。
为解决此问题,我们还提出了 CHEAP (蛋白质压缩沙漏嵌入适应模型),我们学习一个模型来压缩蛋白质序列和结构的联合嵌入。
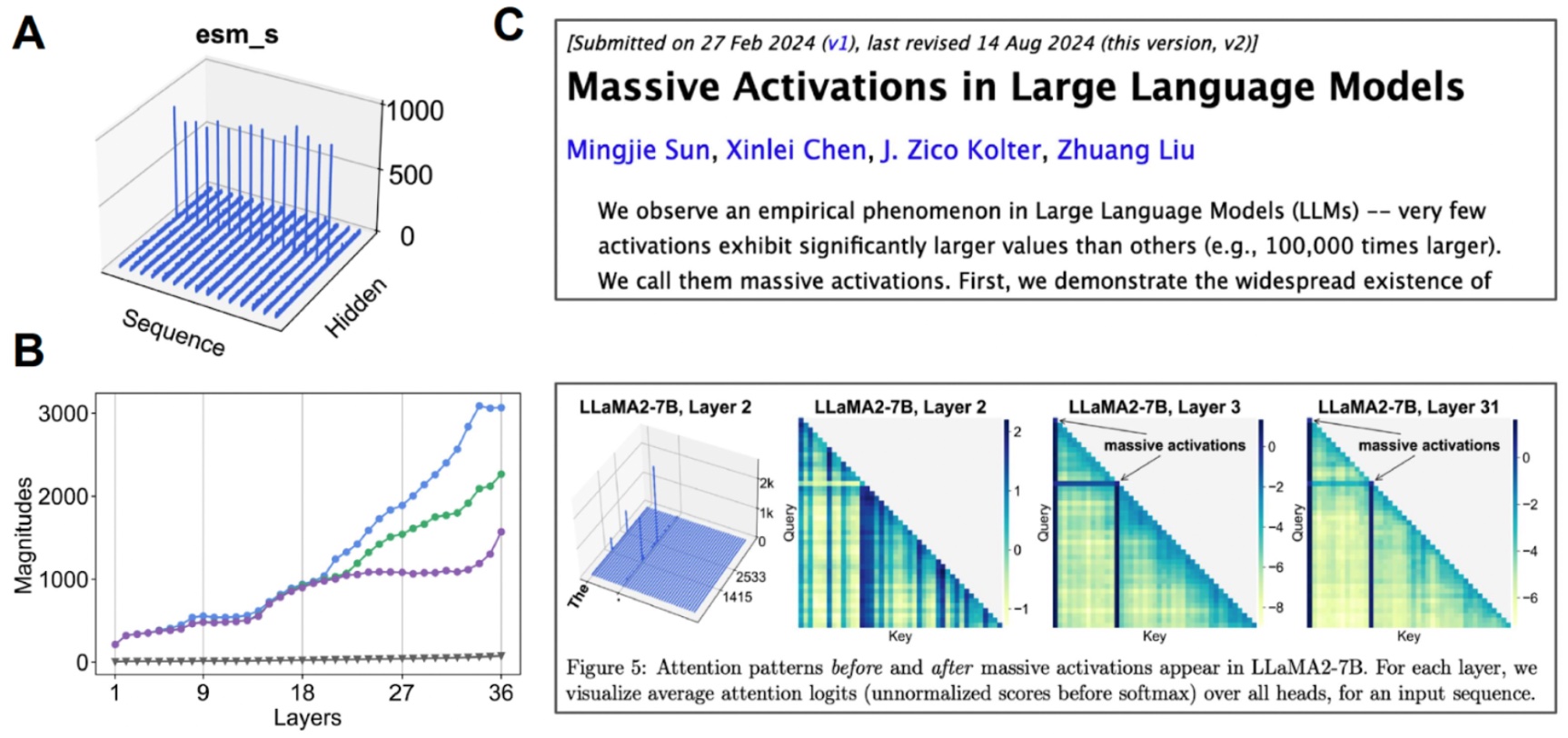
研究潜在空间。 (A) 当我们可视化每个通道的平均值时,一些通道表现出“大规模激活”。(B) 如果我们开始检查与中位数(灰色)相比的前 3 个激活值,我们会发现这种情况发生在许多层中。(C) 对于其他基于 Transformer 的模型也观察到了大规模激活。
我们发现这个潜在空间实际上是高度可压缩的。通过对我们正在使用的基础模型进行一些机械可解释性研究,我们能够创建一个全原子蛋白质生成模型。
接下来是什么?
尽管在这项工作中我们研究了蛋白质序列和结构生成的案例,但我们可以将此方法适用于任何存在从更丰富模态到更稀疏模态的预测器的多模态生成任务。随着蛋白质的序列到结构预测器开始处理越来越复杂的系统(例如 AlphaFold3 也能预测与核酸和分子配体结合的蛋白质),使用相同的方法对更复杂的系统进行多模态生成是很容易想象的。如果您有兴趣合作扩展我们的方法,或在湿实验室中测试我们的方法,请与我们联系!
更多链接
如果您在研究中发现我们的论文很有用,请考虑使用以下 PLAID 和 CHEAP 的 BibTeX:
@article{lu2024generating, title={Generating All-Atom Protein Structure from Sequence-Only Training Data}, author={Lu, Amy X and Yan, Wilson and Robinson, Sarah A and Yang, Kevin K and Gligorijevic, Vladimir and Cho, Kyunghyun and Bonneau, Richard and Abbeel, Pieter and Frey, Nathan}, journal={bioRxiv}, pages={2024--12}, year={2024}, publisher={Cold Spring Harbor Laboratory}
}@article{lu2024tokenized, title={Tokenized and Continuous Embedding Compressions of Protein Sequence and Structure}, author={Lu, Amy X and Yan, Wilson and Yang, Kevin K and Gligorijevic, Vladimir and Cho, Kyunghyun and Abbeel, Pieter and Bonneau, Richard and Frey, Nathan}, journal={bioRxiv}, pages={2024--08}, year={2024}, publisher={Cold Spring Harbor Laboratory}
}您还可以查看我们的预印本(PLAID, CHEAP)和代码库(PLAID, CHEAP)。
一些有趣的蛋白质生成示例!
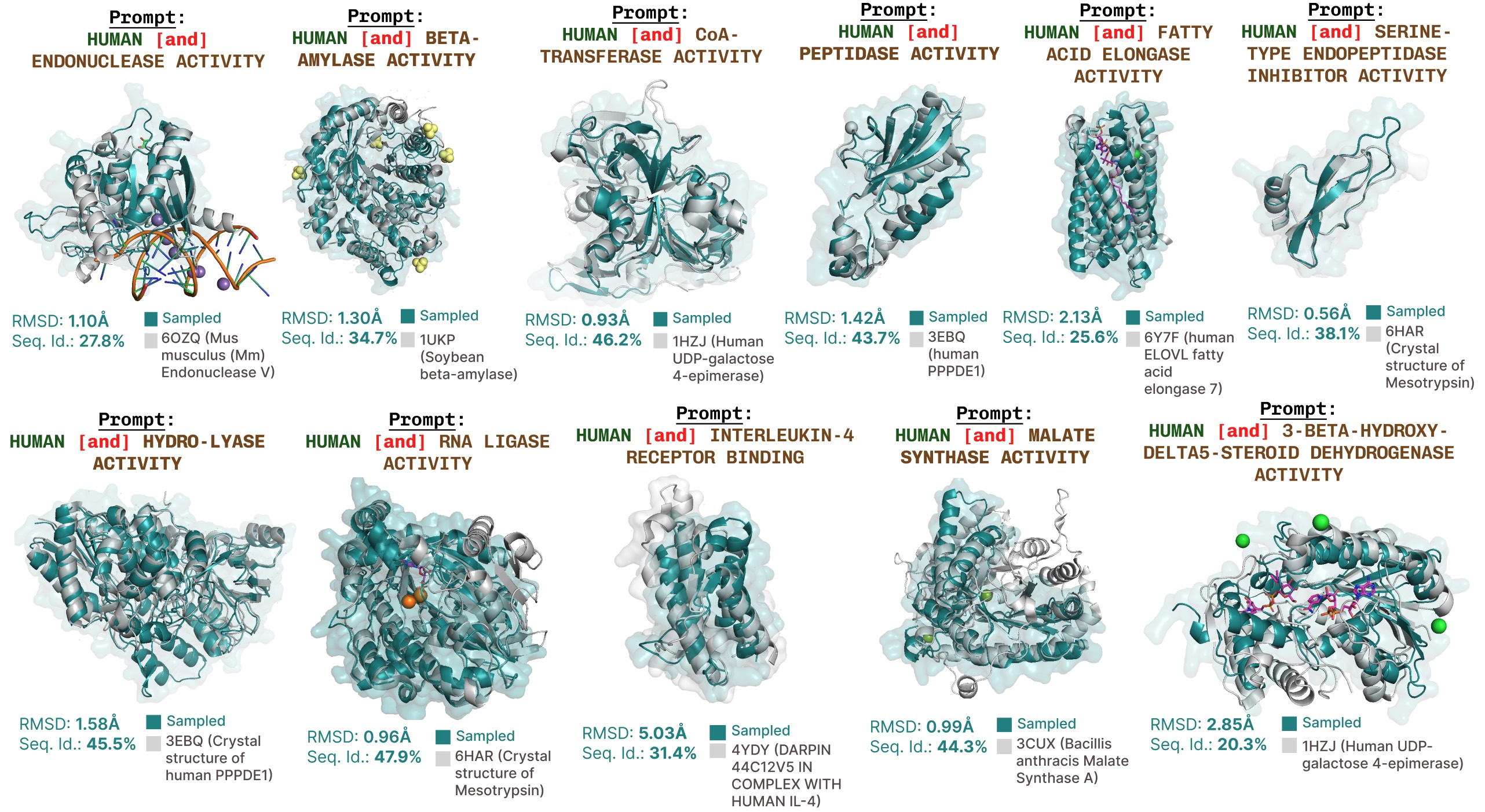
PLAID 通过功能提示生成的附加示例。
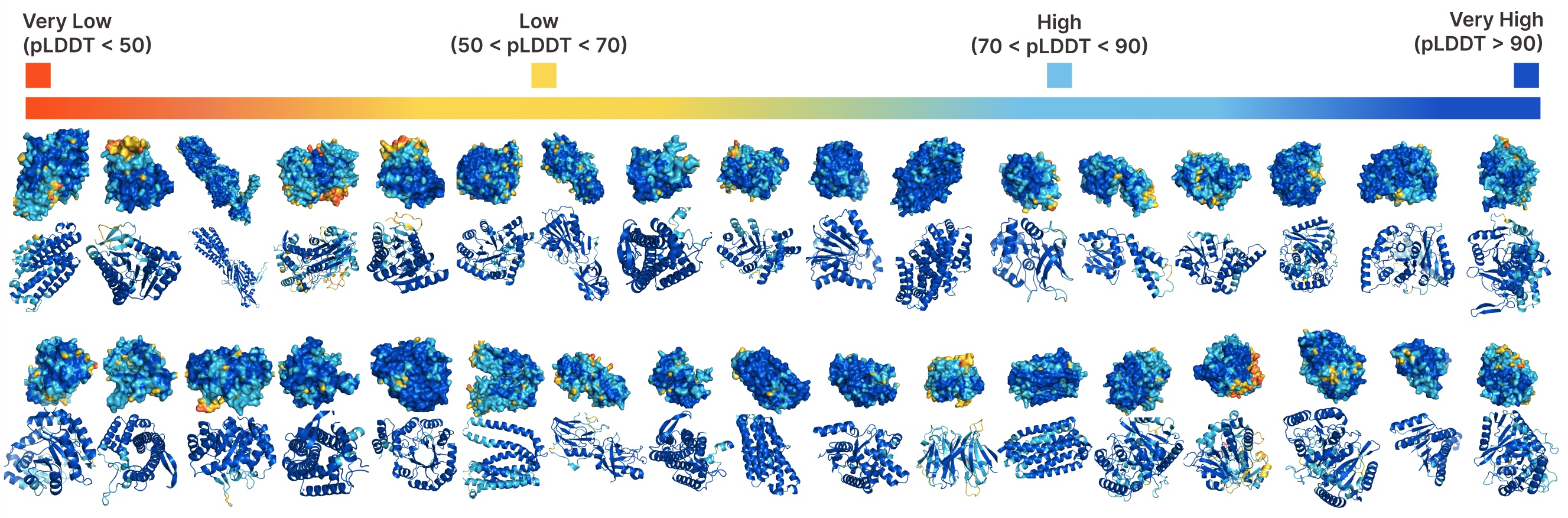
PLAID 的无条件生成。
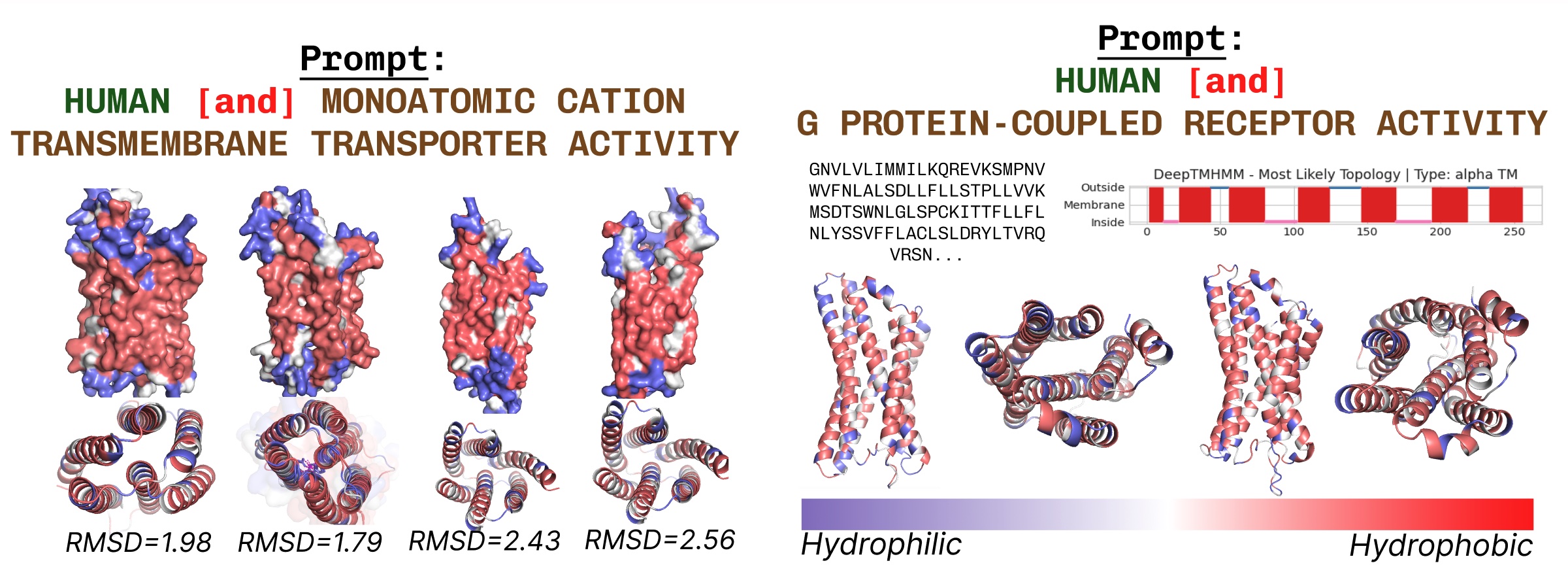
跨膜蛋白在核心位置具有疏水残基,它嵌入脂肪酸层中。当使用跨膜蛋白关键词提示 PLAID 时,这些特征会一致地被观察到。
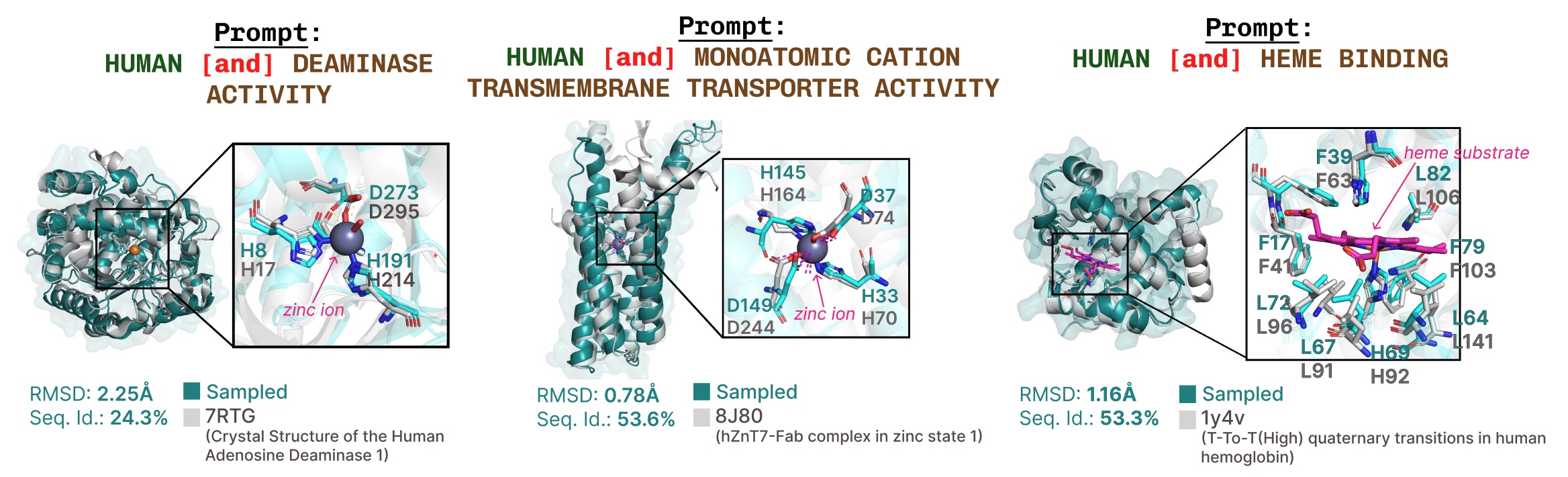
基于功能关键词提示的活性位点再现的附加示例。
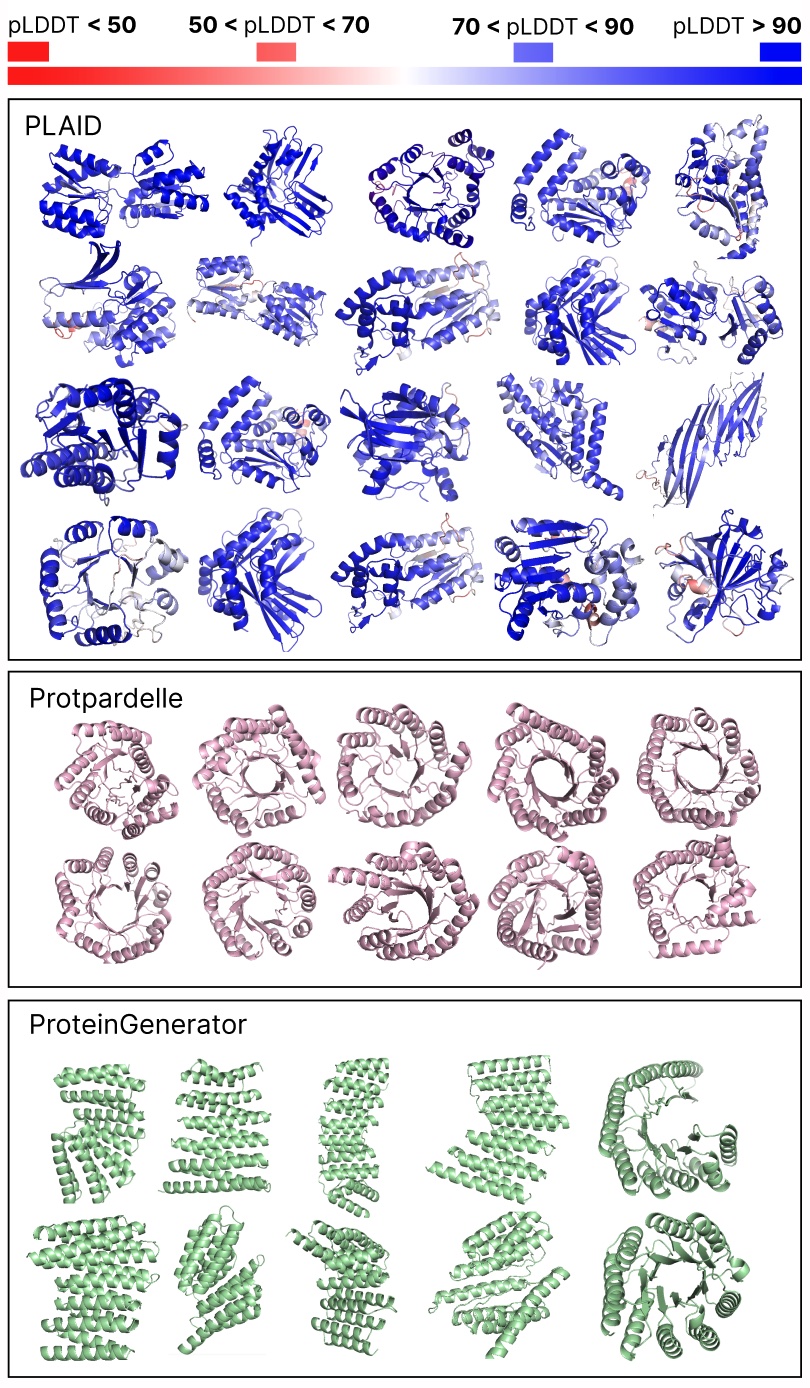
比较 PLAID 与全原子基线的样本。PLAID 的样本具有更好的多样性,并捕获了迄今为止蛋白质生成模型更难学习的 β-折叠模式。
致谢
感谢 Nathan Frey 对本文提出的详细反馈,以及来自 BAIR、基因泰克、微软研究院和纽约大学的合作作者:Wilson Yan、Sarah A. Robinson、Simon Kelow、Kevin K. Yang、Vladimir Gligorijevic、Kyunghyun Cho、Richard Bonneau、Pieter Abbeel 和 Nathan C. Frey。
🚀 想要体验更好更全面的AI调用?
欢迎使用青云聚合API,约为官网价格的十分之一,支持300+全球最新模型,以及全球各种生图生视频模型,无需翻墙高速稳定,文档丰富,小白也可以简单操作。


评论区