📢 转载信息
原文作者:Hasan Poonawala, Kexin Huang, Necibe Ahat, and Pierre de Malliard
本文由 斯坦福大学 Biomni 小组 联合撰写。
生物医学研究人员大约花费 90% 的时间手动处理分散的大量信息。基因泰克(Genentech)在处理 PubMed 上的 3800 万篇生物医学出版物、人类蛋白图谱等公共存储库,以及其跨越数百种疾病的数亿个细胞的内部存储库时遇到的挑战就证明了这一点。专业数据库和分析工具在基因组学、蛋白质组学和病理学等不同模式中迅速激增。研究人员必须跟上庞大的工具格局,留给推动突破性发现的假设驱动型工作的时间就更少。
由基础模型驱动的 AI 智能体提供了一个有希望的解决方案,它可以通过自主规划、执行和适应复杂的科学研究任务。斯坦福大学研究人员构建的 Biomni 就体现了这种潜力。Biomni 是一个通用生物医学 AI 智能体,集成了 150 个专业工具、105 个软件包和 59 个数据库,以执行复杂的分析,例如基因优先排序、药物再利用和罕见病诊断。
然而,将此类智能体部署到生产环境中需要强大的基础设施,该基础设施必须能够处理计算密集型工作流程和多个并发用户,同时保持安全和性能标准。Amazon Bedrock AgentCore 是一套全面的服务,用于使用任何框架或模型部署和运行高能力的智能体,并提供企业级的安全性和可扩展性。
在本文中,我们将向您展示如何使用 AgentCore 实现一个研究智能体,该智能体能够访问 Biomni 提供的 30 多个专业生物医学数据库工具,从而在保持企业级安全性和生产规模的同时加速科学发现。本解决方案的代码可在 Amazon Web Services (AWS) 上生命科学入门智能体开源工具包代码库中找到。分步说明 帮助您部署自己的工具和基础设施,以及 AgentCore 组件和示例。
原型到生产的复杂性差距
将本地生物医学研究原型迁移到可供多个研究团队访问的生产系统,需要解决复杂的基础设施挑战。
具有企业安全性的智能体部署
企业安全挑战包括 OAuth 身份验证、通过可扩展网关安全共享工具、用于研究审计跟踪的全面可观测性,以及处理并发研究工作负载的自动扩展。许多有前景的原型未能投入生产,原因在于在实施这些企业级要求的同时,还要保持准确的生物医学分析所需的专业领域知识的复杂性。
面向会话的研究上下文管理
生物医学研究工作流程通常跨越多个对话,需要在扩展的研究会话中对先前分析、实验参数和研究偏好保持持久的记忆。研究智能体必须保持对正在进行项目的上下文感知,记住特定的蛋白质靶点、实验条件和分析偏好。所有这些都必须在多租户生产环境中促进不同研究人员和研究项目之间的适当会话隔离。
可扩展的工具网关
实施一个可重用的工具网关,该网关能够处理来自研究智能体的并发请求、适当的身份验证和一致的性能,在扩展时变得至关重要。该网关必须使智能体能够通过安全端点发现和使用工具,帮助智能体通过上下文搜索功能找到正确的工具,并在统一的服务中管理入站身份验证(验证智能体身份)和出站身份验证(连接到外部生物医学数据库)。没有这种架构,研究团队将面临阻碍有效扩展的身份验证复杂性和可靠性问题。
解决方案概述
我们使用Strands Agents(一个开源智能体框架)来构建一个研究智能体,该智能体具有用于 PubMed 生物医学文献搜索的本地工具实现。我们通过集成 Biomni 数据库工具扩展了该智能体的能力,提供了对 30 多个专业生物医学数据库的访问权限。
总体架构如下所示。
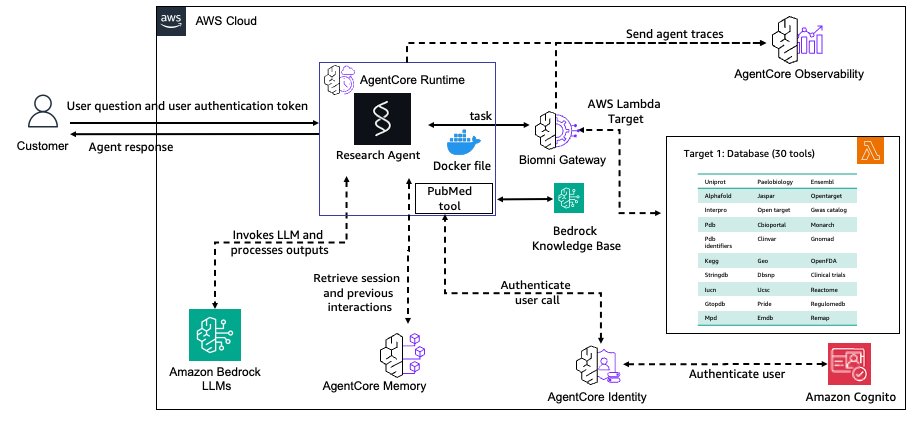
AgentCore Gateway 服务将 Biomni 数据库工具集中化为更安全、可重用的端点,并具有语义搜索功能。AgentCore Memory 服务使用针对研究上下文的专门策略,在整个研究会话中保持上下文感知。安全性由 AgentCore Identity 服务处理,该服务管理用户和工具访问控制的身份验证。部署通过 AgentCore Runtime 服务得到简化,该服务提供可扩展的、托管的部署以及会话隔离。最后,AgentCore Observability 服务支持对研究工作流程进行全面监控和审计,这对科学可再现性至关重要。
步骤 1 – 使用 AgentCore Gateway 创建工具,例如 Biomni 数据库工具
在实际用例中,我们需要将智能体连接到不同的数据源。每个智能体可能会重复相同的工具,导致代码冗余、行为不一致和维护噩梦。AgentCore Gateway 服务通过将工具集中到可重用的安全端点供智能体访问,简化了此过程。结合 AgentCore Identity 服务进行身份验证,AgentCore Gateway 创建了一个企业级的工具共享基础设施。为了给智能体提供更多关于可重用工具的上下文,我们通过注册在网关上的 Biomni 工具 提供了对 30 多个专业公共数据库 API 的访问。该网关通过 Model Context Protocol (MCP) 暴露 Biomni 的数据库工具,允许研究智能体与本地工具(如 PubMed)一起发现和调用这些工具。它处理身份验证、速率限制和错误处理,提供无缝的研究体验。
def create_gateway(gateway_name: str, api_spec: list) -> dict: # JWT authentication with Cognito auth_config = { "customJWTAuthorizer": { "allowedClients": [ get_ssm_parameter(\"/app/researchapp/agentcore/machine_client_id\") ], "discoveryUrl": get_ssm_parameter(\"/app/researchapp/agentcore/cognito_discovery_url\"), } } # Enable semantic search for BioImm tools search_config = {"hcp": {"searchType": "SEMANTIC"}} # Create the gateway gateway = bedrock_agent_client.create_gateway( name=gateway_name, collectionexecution_role_arn, protocolType="MCP", authorizerType="CUSTOM_JWT", authorizerConfiguration=auth_config, protocolConfiguration=search_config, description="My App Template AgentCore Gateway", )api_spec.json) 暴露其可用的工具。
# Gateway Target Configuration lambda_target_config = { "mcp": { "lambda": { "lambdaArn": get_ssm_parameter(\"/app/researchapp/agentcore/lambda_arn\"), "toolSchema": {"inlinePayload": api_spec}, } } } # Create the target create_target_response = gateway_client.create_gateway_target( gatewayIdentifier=gateway_id, name="LambdaUsingSDK", description="Lambda Target using SDK", targetConfiguration=lambda_target_config, credentialProviderConfigurations=[{ "credentialProviderType": "GATEWAY_IAM_ROLE" }], )下表列出了网关上包含的全部 Biomni 数据库工具列表:
| 组 | 工具 | 描述 |
| 蛋白质和结构数据库 | UniProt | 查询 UniProt REST API,获取全面的蛋白质序列和功能信息 |
| AlphaFold | 查询 AlphaFold 数据库 API,获取 AI 预测的蛋白质结构 | |
| InterPro | 查询 InterPro REST API,获取蛋白质结构域、家族和功能位点信息 | |
| PDB (蛋白质数据库) | 查询 RCSB PDB 数据库,获取实验确定的蛋白质结构 | |
| STRING | 查询 STRING 蛋白质相互作用数据库,获取蛋白质-蛋白质相互作用网络 | |
| EMDB (电子显微镜数据银行) | 查询通过电子显微镜确定的三维大分子结构 | |
| 基因组学和变异 | ClinVar | 查询 NCBI 的 ClinVar 数据库,获取临床相关的遗传变异及其解释 |
| dbSNP | 查询 NCBI dbSNP 数据库,获取单核苷酸多态性和遗传变异信息 | |
| gnomAD | 查询 gnomAD,获取群体规模的遗传变异频率和注释信息 | |
| Ensembl | 查询 Ensembl REST API,获取基因组注释、基因信息和比较基因组学数据 | |
| UCSC 基因组浏览器 | 查询 UCSC 基因组浏览器 API,获取基因组数据和注释 | |
| 表达和组学 | GEO (基因表达综合数据库) | 查询 NCBI 的 GEO 数据库,获取 RNA-seq、微阵列和其他基因表达数据集 |
| PRIDE | 查询 PRIDE 数据库,获取蛋白质组学鉴定和质谱数据 | |
| Reactome | 查询 Reactome 数据库,获取生物学通路和分子相互作用信息 | |
| 临床和药物数据 | cBioPortal | 查询 cBioPortal REST API,获取癌症基因组学数据和临床信息 |
| ClinicalTrials.gov | 查询 ClinicalTrials.gov API,获取有关临床研究和试验的信息 | |
| OpenFDA | 查询 OpenFDA API,获取 FDA 药物、设备和食品安全数据 | |
| GtoPdb (药物作用靶点指南) | 查询 Guide to PHARMACOLOGY 数据库,获取药物靶点和药理学数据 | |
| 疾病和表型 | OpenTargets | 查询 OpenTargets Platform API,获取疾病-靶点关联和药物发现数据 |
| Monarch Initiative | 查询 Monarch Initiative API,获取跨物种的表型和疾病信息 | |
| GWAS Catalog | 查询 GWAS Catalog API,获取全基因组关联研究结果 | |
| RegulomeDB | 查询 RegulomeDB 数据库,获取调控变异注释和功能预测信息 | |
| 专业数据库 | JASPAR | 查询 JASPAR REST API,获取转录因子结合位点图谱和基序信息 |
| WoRMS (世界海洋物种名录) | 查询 WoRMS REST API,获取海洋物种分类信息 | |
| Paleobiology Database (PBDB) | 查询 PBDB API,获取化石出现和分类数据 | |
| MPD (小鼠表型数据库) | 查询 Mouse Phenome Database,获取小鼠品系表型数据 | |
| Synapse | 查询 Synapse REST API,获取生物医学数据集和协作研究数据 |
以下是单个工具如何通过 MCP 从我们的测试套件中触发的示例:
# 蛋白质和结构分析 "使用 uniprot 工具查找有关人类胰岛素蛋白的信息" # → 使用蛋白质查询参数触发 uniprot MCP 工具 "使用 alphafold 工具对 uniprot_id P01308 进行结构预测" # → 触发 alphafold MCP 工具进行 3D 结构预测 "使用 pdb 工具查找胰岛素的蛋白质结构" # → 触发 pdb MCP 工具查找晶体结构信息 # 遗传变异分析 "使用 clinvar 工具查找 BRCA1 基因中的致病性变异" # → 使用基因变异参数触发 clinvar MCP 工具 "使用 gnomad 工具查找 BRCA2 变异的群体频率" # → 触发 gnomad MCP 工具获取群体遗传学数据随着工具集的增长,智能体可以使用内置的语义搜索功能,根据任务上下文发现和选择工具。这可以提高智能体性能并降低扩展时的开发复杂性。例如,用户询问:“告诉我关于 HER2 变异 rs1136201 的信息。”语义搜索不会将所有 30 多个工具从网关列出返回给智能体,而是返回‘n’个最相关的工具。例如,Ensembl、Gwas catalog、ClinVar 和 Dbsnp。智能体现在使用更小的工具子集作为模型的输入,以返回更高效、更快速的响应。
下图说明了使用 AgentCore Gateway 进行工具搜索的过程。
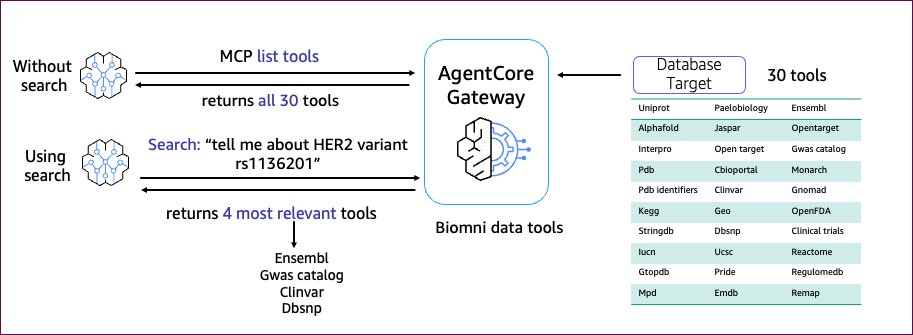
您现在可以使用以下测试脚本测试已部署的 AgentCore 网关,并比较语义搜索如何根据搜索查询缩小相关工具列表的范围:
uv run tests/test_gateway.py --prompt "What tools are available?" uv run tests/test_gateway.py --prompt "Find information about human insulin protein" --use-search步骤 2- 带有本地工具的 Strands 研究智能体
以下代码片段显示了模型初始化,实现了使用 Strands @tool 装饰器声明的 PubMed 本地工具。我们在 research_tools.py 中实现了 PubMed 工具,该工具调用 PubMed API,使智能体能够在执行上下文中进行生物医学文献搜索。
from agent.agent_config.tools.PubMed import PubMed @tool( name="Query_pubmed", description=("Query PubMed for relevant biomedical literature based on the user's query. " "This tool searches PubMed abstracts and returns relevant studies with " "titles, links, and summaries."), ) def query_pubmed(query: str) -> str: """ Query PubMed for relevant biomedical literature based on the user's query. This tool searches PubMed abstracts and returns relevant studies with titles, links, and summaries. Args: query: The search query for PubMed literature Returns: str: Formatted results from PubMed search """ pubmed = PubMed() print(f"\nPubMed Query: {query}\n") result = pubmed.run(query) print(f"\nPubMed Results: {result}\n") return result-
-
- 创建 Strands 研究智能体,其中包含本地工具和Claude Sonnet 4 交错思考。
-
class ResearchAgent: def __init__( self, bearer_token: str, memory_hook: MemoryHook = None, session_manager: AgentCoreMemorySessionManager = None, bedrock_model_id: str = "us.anthropic.claude-sonnet-4-20250514-v1.0", #bedrock_model_id: str = "openai.gpt-oss-120b-1.0", # Alternative system_prompt: str = None, tools: List[callable] = None, ): self.model_id = bedrock_model_id # For Anthropic Sonnet 4 interleaved thinking self.model = BedrockModel( model_id=self.model_id, additional_request_fields={ "anthropic_beta": ["interleaved-thinking-2025-05-14"], "thinking": {"type": "enabled", "budget_tokens": 8000}, }, ) self.system_prompt = ( system_prompt if system_prompt else """ You are a **Comprehensive Biomedical Research Agent** specialized in conducting systematic literature reviews and multi-database analyses to answer complex biomedical research questions. Your primary mission is to synthesize evidence from both published literature (PubMed) and real-time database queries to provide comprehensive, evidence-based insights for pharmaceutical research, drug discovery, and clinical decision-making. Your core capabilities include literature analysis and extracting data from 30+ specialized biomedical databases** through the Bioimm gateway, enabling comprehensive data analysis. The database tool categories include genomics and genetics, protein structure and function, pathways and system biology, clinical and pharmacological data, expression and omics data and other specialized databases. """ )- 此外,我们实施了引用功能,它使用结构化的系统提示来强制执行带编号的文内引用 [1]、[2]、[3] 以及标准化的参考格式,用于学术文献和数据库查询,确保每条数据源都得到适当的归属。这使得研究人员能够快速访问和引用支持其生物医学研究查询和发现的科学文献。
""" <citation_requirements> - ALWAYS use numbered in-text citations [1], [2], [3], etc. when referencing any data source - Provide a numbered "References" section at the end with full source details - For academic literature: format as "1. Author et al. Title. Journal. Year. ID: [PMID/DOI], available at: [URL]" - For database sources: format as "1. Database Name (Tool: tool_name), Query: [query_description], Retrieved: [current_date]" - Use numbered in-text citations throughout your response to support all claims and data points - Each tool query and each literature source must be cited with its own unique reference number - When tools return academic papers, cite them using the academic format with full bibliographic details - Structure: Format each reference on a separate line with proper numbering - NO bullet points - Present the References section as a clean numbered list, not a confusing paragraph - Maintain sequential numbering across all reference types in a single "References" section </citation_requirements> """ 您现在可以本地测试您的智能体:
uv run tests/test_agent_locally.py --prompt "Find information about human insulin protein" uv run tests/test_agent_locally.py --prompt "Find information about human insulin protein" --use-search步骤 3 - 添加持久内存以提供上下文研究协助
研究智能体实现了 AgentCore Memory 服务,其中包含三种策略:用于事实研究上下文的语义策略,用于研究方法的用户偏好策略,以及用于会话连续性的摘要策略。AgentCore Memory 会话管理器与 Strands 会话管理集成,在查询前检索相关上下文并在响应后保存交互。这使得智能体能够在会话中记住研究偏好、正在进行的项目和领域专业知识,而无需手动重新建立上下文。
# 使用研究对话测试内存功能
python tests/test_memory.py load-conversation<br />python tests/test_memory.py load-prompt "My preferred response format is detailed explanations"步骤 4 - 使用 AgentCore Runtime 进行部署
为了部署我们的智能体,我们使用 AgentCore Runtime 来配置和启动研究智能体作为托管服务。部署过程使用智能体的主要入口点(agent/main.py)配置运行时,分配 IAM 执行角色以供 AWS 服务访问,并支持 OAuth 和 IAM 身份验证模式。部署后,运行时成为一个可扩展的无服务器智能体,可以通过 API 调用调用。智能体自动处理会话管理、内存持久化和工具编排,同时提供对 Biomni 网关和本地研究工具的安全访问。
agentcore configure --entrypoint agent/main.py -er arn:aws:iam::<Account-Id>:role/<Role> --name researchapp<AgentName>有关使用 AgentCore Runtime 进行部署的更多信息,请参阅Amazon Bedrock AgentCore 开发者指南中的AgentCore Runtime 入门。
智能体实战
以下是展示智能体在不同领域(药物作用机制分析、遗传变异研究和通路探索)能力的三个典型研究场景:对于每个查询,智能体都会自主确定要使用哪些工具组合、制定适当的子查询、分析返回的数据,并以适当的引用合成一份全面的研究报告。随附的演示视频展示了完整的智能体工作流程,包括工具选择、推理和响应生成。
- 对曲妥珠单抗(赫赛汀)的作用机制和耐药机制进行全面分析,您将需要:
- HER2 蛋白质结构和结合位点
- 受影响的下游信号通路
- 来自临床数据的已知耐药机制
- 当前研究联合疗法的临床试验
- 预测治疗反应的生物标志物查询相关数据库以提供全面的研究报告。
- 分析 BRCA1 变异在乳腺癌风险和治疗反应中的临床意义。研究:
- 致病性 BRCA1 变异的群体频率
- 临床意义和致病性分类
- 相关癌症风险和外显率估计
- 治疗影响(PARP 抑制剂、铂类药物)
- BRCA1 阳性患者的当前临床试验使用多个数据库提供全面证据
以下视频是生物医学研究智能体的演示:
可扩展性和可观测性
部署复杂 AI 智能体时最关键的挑战之一是确保它们能够可靠地扩展,同时对其操作保持全面的可见性。生物医学研究工作流程本质上是不可预测的——单个基因组分析可能会处理数千个文件,而文献综述可能涉及数百万篇出版物。传统基础设施难以应对这些动态工作负载,特别是在处理需要严格隔离不同研究项目敏感研究数据时。在此部署中,我们使用 Amazon Bedrock AgentCore Observability 来可视化智能体工作流程的每一步。您可以使用此服务检查智能体的执行路径、审计中间输出,并调试性能瓶颈和故障。对于生物医学研究而言,这种程度的透明度不仅仅是有帮助的——它对于监管合规和科学可再现性至关重要。
会话(Sessions)、跟踪(traces)和跨度(spans)在可观测性框架中形成了三层层次关系。一个会话包含多个跟踪,每个跟踪代表在更广泛会话上下文中的一次离散交互。每个跟踪包含多个捕获细粒度操作的跨度。下图显示了一个智能体的用量:生产环境中的会话数量、令牌使用量和错误率
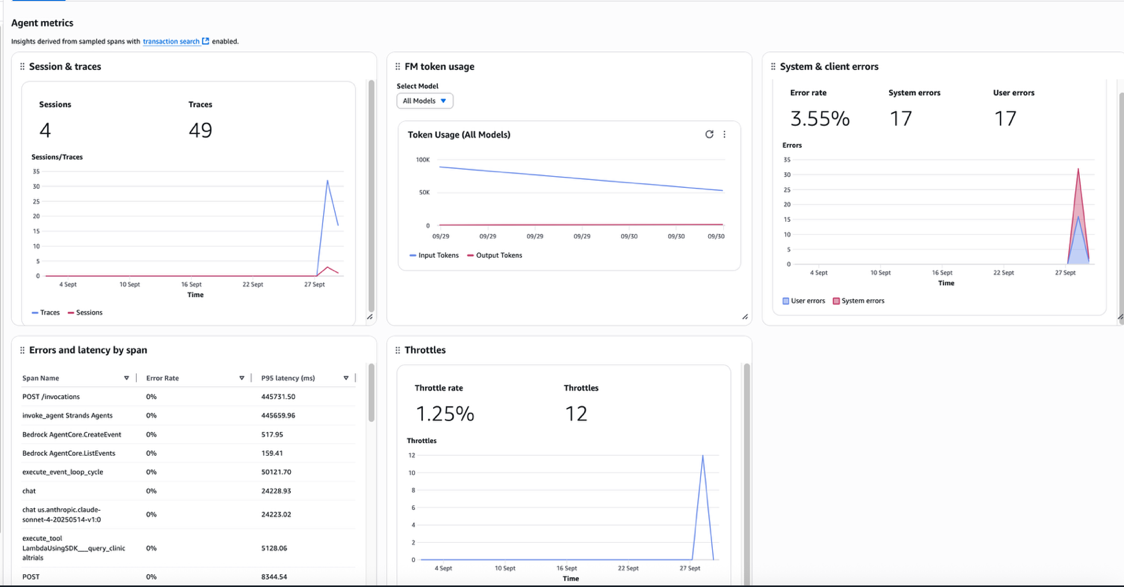
下图显示了生产中的智能体及其使用情况(会话数、调用次数)
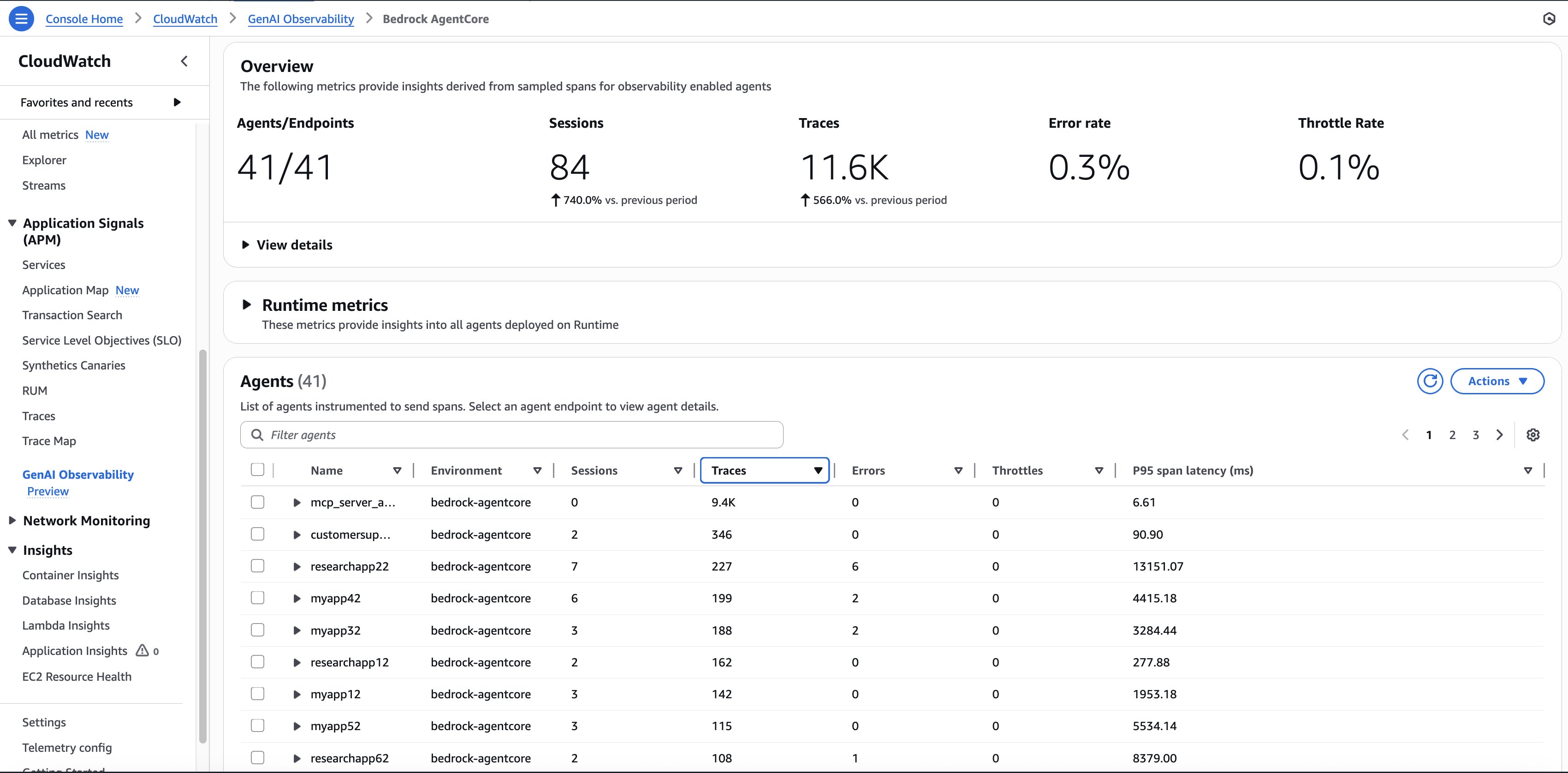
内置的仪表板显示了性能瓶颈,并确定了某些交互可能失败的原因,从而能够持续改进,并减少平均检测时间 (MTTD) 和平均修复时间 (MTTR)。对于可能延迟关键研究时间表的生物医学应用,这种快速问题解决能力确保了研究势头的持续。
未来方向
尽管本次实施侧重于一小部分工具,但 AgentCore Gateway 架构在设计上具有可扩展性。研究团队可以通过使用 MCP 协议,在无需代码更改的情况下无缝添加新工具。新注册的工具可被智能体自动发现,从而使您的研究基础设施能够与快速变化的工具集同步发展。
对于需要代码执行的计算分析,... [内容被截断]


评论区